In dem Profilbereich geht es darum, Prozesse des Lebens zu verstehen, um neuartige Therapien und effiziente biotechnologische Anwendungen zu entwickeln und dynamische Zusammenhänge in Ökosystemen zu beschreiben. Dazu werden molekulare Prozesse, zelluläre Architekturen und das Verhalten von Organismen experimentell und theoretisch mit modernen Methoden aus allen Natur- und Lebenswissenschaften untersucht, die zu einem großen Teil in Frankfurt entwickelt wurden. Die Forschungsschwerpunkte umfassen RNA-basierte Prozesse, Membran- und Organelldynamiken, neuronale molekulare und zelluläre Architekturen, zelluläre und Zell-Zell-Interaktionen und lichtgesteuerte Prozesse.
Vielen Krankheiten liegen äußerst komplexe Prozesse zugrunde. Wie interagieren zum Beispiel Viren und Bakterien mit dem menschlichen Körper? Wie entwickeln sich Entzündungsreaktionen? Was, wenn das zelluläre Gleichgewicht kippt? Das Clusterprojekt ENABLE erforscht diese Prozesse, um neue therapeutische Strategien zu entwickeln.
Damit der menschliche Körper funktionieren und erfolgreich schädliche Einflüsse abwehren kann, ist ein gewaltiges Kommunikationsnetzwerk aktiv. So gibt es in Zellen viele verschiedenen Mechanismen der Qualitätskontrolle, die das innere Gleichgewicht – Wissenschaftler*innen sprechen auch von Homöostase – aufrechterhalten. Dazu zählt zum Beispiel, dass Zellen mögliche Schäden aufspüren und reparieren können, dass sie überflüssige Bestandteile entsorgen und fehlregulierte Signalwege wieder unter Kontrolle bringen können.
Gelingt es den Zellen nicht, ihr Gleichgewicht zu erhalten, so sind die Folgen gravierend: Fehlende Kontrolle der Zellteilung etwa ist eine Ursache für Krebs. Überreagierende Signalwege können chronische Entzündungsprozesse auslösen. Und falsch gefaltete Proteine, die sich in Zellen anreichern, spielen eine zentrale Rolle bei neurodegenerativen Erkrankungen wie Alzheimer-Demenz, Morbus Parkinson und Amyotropher Lateralsklerose (ALS).
Wie überwacht nun die Zelle ihr Gleichgewicht? Wie reagieren Körperzellen auf Viren und Bakterien? Und wie entstehen Entzündungen auf zellulärer und molekularer Ebene, was befeuert sie und wie werden sie unter Kontrolle gebracht? Dies sind Fragen, denen das Clusterprojekt ENABLE (Unravelling mechanisms driving cellular homeostasis, inflammation and infection to enable new approaches in translational medicine) in drei komplementären Forschungsschwerpunkten nachgeht.
Um Ansätze für neuartige Medikamente zu finden, interessieren sich die ENABLE-Forscher*innen besonders für das komplexe Zusammenspiel der Signalwege, mit denen Zellen ihre Homöostase überwache, immunologische Antworten steuern und Bedrohungen abwehren.
Zu den zentralen Mechanismen der Qualitätskontrolle zählen das Ubiquitin-Proteasom-System und der Autophagie-Lysosom-Stoffwechselweg – zwei Systeme, die dem Abbau beschädigter und nicht mehr benötigter Zellbestandstandteile dienen. ENABLE-Sprecherin Prof. Maike Windbergs vom Institut für Pharmazeutische Technologie der Goethe-Universität erklärt: »Wir möchten in bislang ungekannter Präzision die molekulare und räumliche Organisation der zugrundeliegenden Prozesse untersuchen und vor allem verstehen, wie die verschiedenen Signalnetzwerke in der Zelle übergeordnet koordiniert werden. Denn es gibt ganz offensichtlich Verbindungen zwischen Entzündungsreaktionen und den Systemen der zellulären Qualitätskontrolle. Das ist hoch spannend, weil sich daraus neue Möglichkeiten zur Modulation von zellulären Antworten ergeben.“
In einem zweiten großen Themenblock untersuchen ENABLE-Wissenschaftler*innen, wie Bakterien und Viren mit Körperzellen interagieren, welche Immunantworten hierdurch ausgelöst werden und wie es in der Folge zu Gewebeschäden und Krankheiten kommt. Denn so wie das Spike-Protein von SARS-CoV-2 erst die Infektion der menschlichen Wirtszellen ermöglicht, besitzen Viren und Bakterien jeweils sehr spezifische Moleküle, mit deren Hilfe sie in Zellen oder Gewebe des Wirts eindringen oder sich dem Zugriff des Immunsystems entziehen. »Nur wenn wir diese Mechanismen genau verstehen, können wir wirksame Medikamente etwa gegen Antibiotika-resistente Bakterien oder neue Viren wie SARS-CoV-2 entwickeln«, sagt ENABLE-Sprecher Prof. Ivan Đikić vom Institut für Biochemie II der Goethe-Universität. Ziel ist insbesondere, breit anwendbare, schnell anpassbare Konzepte zur Bekämpfung bestehender und neu auftretender Bedrohungen zu entwickeln.
„Heute wissen wir, dass Entzündungen komplexe Krankheiten verursachen und befördern können.“
Maike Windbergs
Entzündungsreaktionen schließlich stehen im Fokus des dritten ENABLE-Themenblocks. Klassischerweise wurden Entzündungen lediglich als Schutzreaktion des Körpers gegenüber Verletzungen und eindringenden Keimen betrachtet: Beschädigte oder von Viren befallene Zellen senden Signalstoffe aus, die wiederum Zellen des Immunsystems anlocken, diese aktivieren und molekulare Verteidigungsmechanismen in Gang setzen. »Heute wissen wir«, so Windbergs, »dass Entzündungen und die entsprechenden Antworten des Immunsystems auch komplexe Krankheiten wie Krebs, Herzerkrankungen und Morbus Alzheimer verursachen und befördern können. Indem wir entschlüsseln, wie Entzündungen auf zellulärer und molekularer Ebene entstehen, sich entwickeln und wieder aufgelöst werden, legen wir die Basis für die Entwicklung neuer therapeutischer Strategien. Unser Ziel sind zukunftsträchtige Ansätze für Entzündungstherapien, die stärker auf den individuellen Patienten zugeschnitten werden können.«
Alle drei ENABLE-Themen – Homöostase, Infektionen und Entzündungen – greifen über zelluläre Signalketten eng ineinander. Dazu Đikić: »Daher arbeiten wir interdisziplinär zusammen und setzen modernste Technologien sowie neuartige, chemische und biologische Werkzeuge ein, die es uns erlauben, zelluläre Funktionen mit bislang ungekannter Präzision zu analysieren.« Innovative Plattform-Technologien sind ein zentraler Bestandteil von ENABLE und entscheidend für den Erfolg der ambitionierten Forschungsprojekte. Substanzielle Unterstützung erhält der Ausbau solcher Technologien auch durch das Zukunftscluster PROXIDRUGS, das sich 2021 in einem Bundeswettbewerb durchgesetzt hat und mit bis zu 15 Millionen Euro gefördert wird. Die neuartige Substanzklasse der durch Nähe induzierten Wirkstoffe (»proximity-inducing drugs«) erlaubt den gezielten Abbau von krankheitsrelevanten Proteinen und eröffnet damit neue Therapieoptionen bei diversen Erkrankungen.
Das Clusterprojekt ENABLE baut auf bereits existierenden starken Forschungsverbünden und Technologieplattformen an der Goethe-Universität, dem Max-Plack-Institute für Biophysik, dem Frankfurt Institute for Advanced Studies, dem Georg-Speyer-Haus und dem Fraunhofer-Institut für Translationale Medizin und Pharmakologie auf. Für die Zukunft wird das Netzwerk noch größer gespannt: Für die Initiative EMTHERA (Emerging Therapeutics) hat ENABLE eine schlagkräftige Allianz mit Kolleg*innen der Johannes Gutenberg-Universität Mainz geschmiedet. An beiden Standorten arbeiten zahlreiche Fachbereiche – von der Medizin über die Chemie und die Biowissenschaften bis hin zu den Wirtschaftswissenschaften – interdisziplinär zusammen.
(mbe/pb)
ENABLE
Goethe-Universität (Sprecher), Max-Planck-Institut für Biophysik, Frankfurt Institute for Advanced Studies, Georg-Speyer-Haus und Fraunhofer-Institut für Translationale Medizin und Pharmakologie
Beteiligte Institutionen-Partner:
Fachbereich Wirtschaftswissenschaften, Informatik/Mathematik, Biochemie/Chemie/Pharmazie, Biowissenschaften und Medizin, Max-Delbrück-Centrum (Berlin), Max Planck Institute for Heart and Lung Research (Bad Nauheim), Max Planck Institute of Molecular Cell Biology and Genetics (Dresden)
Netzwerken für neue Therapien


Nachgefragt bei...
Maike Windbergs und Ivan Đikić
Welches Problem wollen Sie mit ENABLE gerne besser verstehen?
Maike Windbergs: Durch ENABLE möchten wir die Schnittstellen von Infektionen, Entzündungen und Immunantworten besser verstehen lernen, um neue Ansätze für bisher nicht adäquat heilbare Krankheiten zu entwickeln.
Ivan Đikić: COVID-19 ist hier ein gutes Beispiel – schwere Verläufe gehen meist mit einer überschießenden Immunreaktion einher, bei der das Immunsystem letztendlich körpereigenes Gewebe angreift und zerstört.
Ivan Đikić: COVID-19 ist hier ein gutes Beispiel – schwere Verläufe gehen meist mit einer überschießenden Immunreaktion einher, bei der das Immunsystem letztendlich körpereigenes Gewebe angreift und zerstört.
Was ist Ihnen daran persönlich wichtig?
Windbergs: Auch wenn wir beide in der Grundlagenforschung arbeiten, wurde unsere Ausbildung als Mediziner beziehungsweise Apothekerin von der Motivation geleitet, einige der größten biomedizinischen Herausforderung unserer Zeit zu lösen. Dies kann nur in einem interdisziplinären Konsortium erreicht werden, weshalb es wichtig ist, ein Team aus verschiedenen Spezialistinnen und Spezialisten zusammenzustellen, um völlig neue Forschungsansätze zu finden.
Đikić: Dieses Projekt ermöglicht uns, die bereits vorhandenen Plattform-Technologien weiter auszubauen und so einzusetzen, dass neue Erkenntnisse zügig in den klinischen Kontext transferiert werden können. Abgesehen davon, dass wir damit einen wichtigen Beitrag für neue therapeutische Strategien leisten, tragen wir auch entscheidend zur Ausbildung einer neuen Generation von interdisziplinären Forschern und Forscherinnen für die Zukunft bei.
Đikić: Dieses Projekt ermöglicht uns, die bereits vorhandenen Plattform-Technologien weiter auszubauen und so einzusetzen, dass neue Erkenntnisse zügig in den klinischen Kontext transferiert werden können. Abgesehen davon, dass wir damit einen wichtigen Beitrag für neue therapeutische Strategien leisten, tragen wir auch entscheidend zur Ausbildung einer neuen Generation von interdisziplinären Forschern und Forscherinnen für die Zukunft bei.
Was ist die größte Hürde?
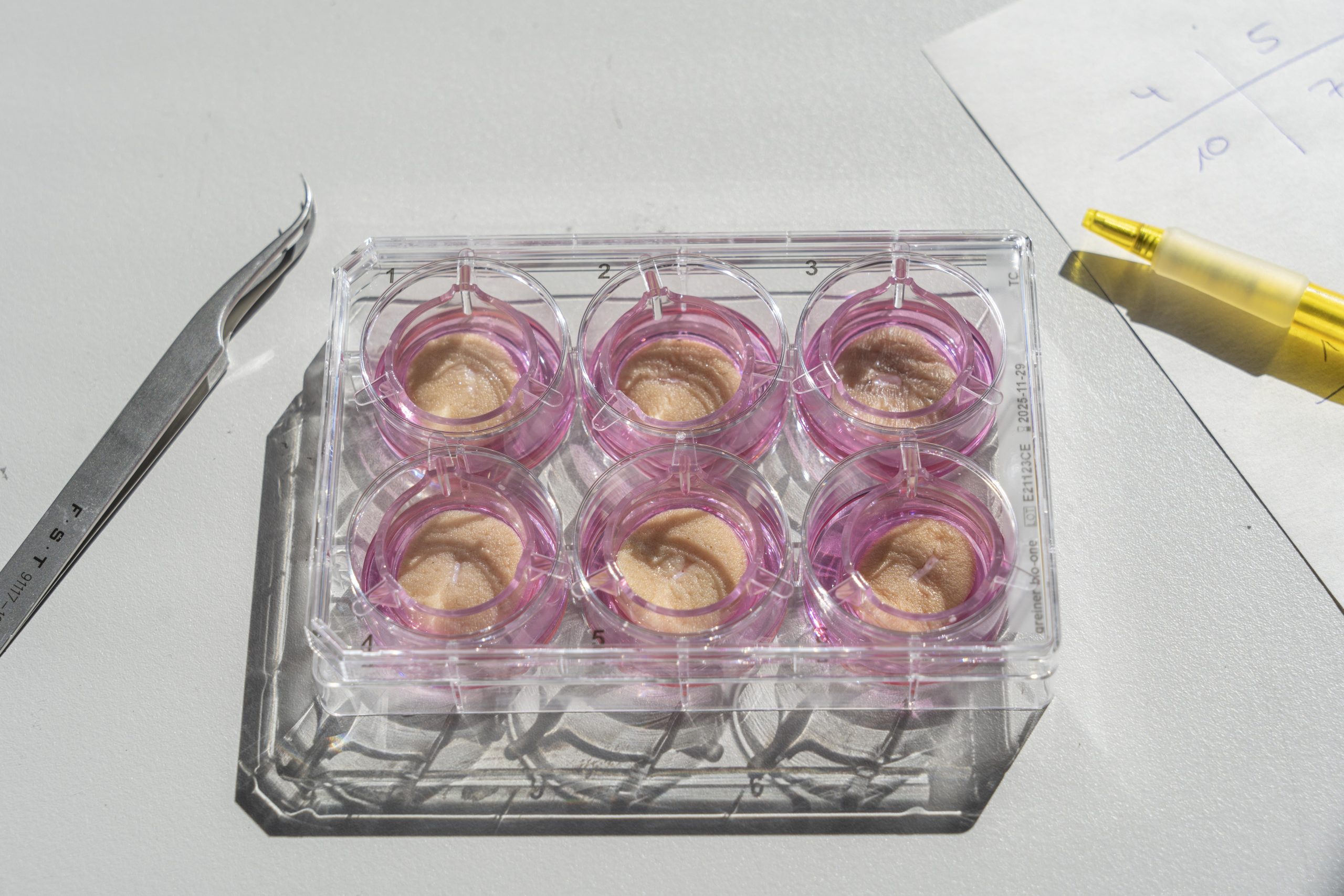
Windbergs: Eine große Herausforderung besteht natürlich darin, die Schnittstellen und das Zusammenspiel zwischen verschiedenen Mechanismen im menschlichen Körper zu verstehen und dafür die geeigneten Tools und Modelle – zum Beispiel humane 3D-Gewebemodelle – zu entwickeln. Mit solchen Modellen ahmen wir die Situation im menschlichen Körper gezielt nach, um dann die Übertragbarkeit in die Klinik zu ermöglichen. Auf diese Weise können wir zum Beispiel zielgerichtet Strategien für hochwirksame Antibiotika und Virostatika entwickeln.
Đikić: Am Ende kennen wir aber nie alle Hürden, die in einem Projekt auf uns zukommen werden. Das ist ja in gewisser Weise auch der besondere wissenschaftliche Reiz – wir bewegen uns auf neuem Terrain, und da gibt es immer wieder überraschende Entdeckungen und ebenso unerwartete Herausforderungen. Es ist daher essenziell, über das richtige Team und die geeignete Infrastruktur zu verfügen, um flexibel und schnell auf neue Erkenntnisse reagieren zu können.
Đikić: Am Ende kennen wir aber nie alle Hürden, die in einem Projekt auf uns zukommen werden. Das ist ja in gewisser Weise auch der besondere wissenschaftliche Reiz – wir bewegen uns auf neuem Terrain, und da gibt es immer wieder überraschende Entdeckungen und ebenso unerwartete Herausforderungen. Es ist daher essenziell, über das richtige Team und die geeignete Infrastruktur zu verfügen, um flexibel und schnell auf neue Erkenntnisse reagieren zu können.
Gibt es eine Erkenntnis, die Sie besonders geprägt hat?
Windbergs: Eine prägende Erfahrung für uns alle war sicherlich, wie die in Mainz entwickelte Plattform-Technologie zur Produktion mRNA-basierter Impfstoffe in Rekordzeit zur Entwicklung wirksamer COVID-19-Impfstoffe geführt hat. Aus meinem Labor nehme ich insbesondere die gelungene Nachbildung von lebensfähigen menschlichen 3D-Geweben mit, mit denen wir auch Krankheitszustände simulieren können. Diese werden für die geplanten Projekte äußerst wertvoll sein, um im Gewebekontext Mechanismen zu untersuchen und therapeutische Strategien zu validieren.
Đikić: Vor einigen Jahren hat mein Labor eine bahnbrechende Entdeckung gemacht, als wir eine bislang unbekannte Art der Ubiquitin-Modifikation durch Legionellen-Enzyme mechanistisch bis ins Detail aufklären konnten. Der Einsatz eines ganzen Repertoires aus struktur- und funktionsaufklärenden Studien ermöglichte es uns, sowohl molekulare Details als auch die Abläufe im zellulären Kontext einer Legionellen-Infektion zu verstehen. Dieses breite Methodenspektrum, das wir mittlerweile auch mit bioinformatischen Methoden, zum Beispiel dynamischer Modellierung, kombinieren, ist zum unverzichtbaren Rüstzeug für die Zukunft geworden. Mit unserem Zukunftscluster PROXIDRUGS haben wir zudem erstmals die Grundlage für die Entwicklung von Molekülen gelegt, die auch therapeutisch einsetzbar wären.
Đikić: Vor einigen Jahren hat mein Labor eine bahnbrechende Entdeckung gemacht, als wir eine bislang unbekannte Art der Ubiquitin-Modifikation durch Legionellen-Enzyme mechanistisch bis ins Detail aufklären konnten. Der Einsatz eines ganzen Repertoires aus struktur- und funktionsaufklärenden Studien ermöglichte es uns, sowohl molekulare Details als auch die Abläufe im zellulären Kontext einer Legionellen-Infektion zu verstehen. Dieses breite Methodenspektrum, das wir mittlerweile auch mit bioinformatischen Methoden, zum Beispiel dynamischer Modellierung, kombinieren, ist zum unverzichtbaren Rüstzeug für die Zukunft geworden. Mit unserem Zukunftscluster PROXIDRUGS haben wir zudem erstmals die Grundlage für die Entwicklung von Molekülen gelegt, die auch therapeutisch einsetzbar wären.
Prof. Dr. Maike Windbergs und Prof. Dr. Ivan Đikić sind Sprecher des Forschungsprojekts ENABLE. Die Pharmazeutin Windbergs ist Inhaberin der Tierschutz-Professur am Institut für Pharmazeutische Technologie, der Molekularbiologe Đikić ist Direktor des Instituts für Biochemie II.
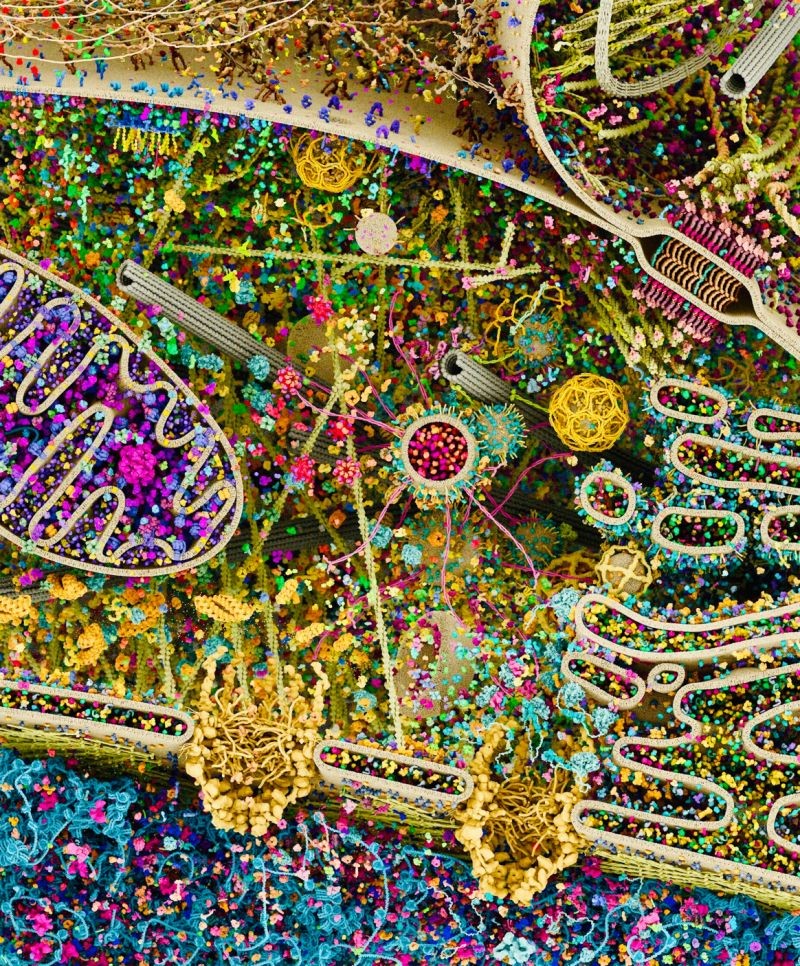
Bis auf die Mikroebene der Atome können Zellen heute erforscht werden – dank moderner bildgebender Verfahren. Im Forschungsprojekt SCALE sollen mithilfe jüngster technologischer Fortschritte Krankheiten besser verstanden werden.
Die Strukturen von RNA und Proteinkomplexen aufzuklären, darin liegt traditionell eine große Stärke der Naturwissenschaftler*innen auf dem Campus Riedberg. »Daran wollen wir mit SCALE anknüpfen«, sagt die Biologin Michaela Müller-McNicoll, die gemeinsam mit dem Biophysiker Achilleas Frangakis Sprecher*in des interdisziplinären Clusters SCALE (»Subcellular Architecture of Life«) ist. Das Ziel des Clusterprojekts ist es, genauer hinzuschauen auf die Vorgänge, die in einem lebenden Organismus innerhalb der Zellen ablaufen. Und zwar so genau, dass mit modernsten bildgebenden Verfahren einzelne Moleküle und sogar einzelne Atome sichtbar gemacht werden. „Damit wollen wir untersuchen, wie Moleküle untereinander und mit ihrer Umgebung in einer dicht gepackten Zelle interagieren“, so Müller-McNicoll.
„Wir untersuchen, wie Moleküle in einer dicht gepackten Zelle interagieren.“
Michaela Müller-McNicoll
»Eine Zelle kann man sich vorstellen wie ein Haus«, erläutert sie, »genauso wie das Haus eine Küche, ein Badezimmer, ein Wohn- und ein Schlafzimmer und vielleicht eine Abstellkammer hat, so besitzt auch die Zelle verschiedene Abteilungen oder Organellen: Zellkern, Mitochondrien, Ribosomen und Synapsen, also Kontakte zwischen Nervenzellen.« Die Zimmer sind von Wänden umgeben, durch Türen oder Fenster können Dinge hinein- und heraustransportiert werden. »Und genauso, wie man einen Raumteiler verschieben und einen Tisch von einer Ecke in die andere stellen kann, besitzt die Zelle zusätzlich auch dynamische Architekturelemente, die sich bilden oder unter veränderten Umständen auch wieder abbauen«, fährt die Biologin fort. Dazu gehören auch Synapsen, also Kontakte von Nervenzellen zu anderen Nervenzellen. Müller-McNicoll, Frangakis und die anderen an SCALE beteiligten Forschenden wollen daher zunächst auflisten, welche Bauelemente vorhanden sind, dann beobachten, wie sich der Bestand verändert, und herausfinden, wie diese sich zu größeren funktionellen Architekturelementen zusammensetzen. Langfristig soll es um die Frage gehen, wie der dynamische Gleichgewichtszustand einer lebenden Zelle von diesen Veränderungen beeinflusst wird: Ob eine Zelle unter Stress mit »architektonischen Veränderungen« reagiert und wie sich diese auswirken – in der einzelnen Zelle und im umgebenden Gewebe.
Müller-McNicoll nennt drei Ziele, die durch die Arbeit an SCALE erreicht werden sollen: »Zum einen wollen wir sogenannte dynamische Modelle erstellen: also alle Bestandteile der Zelle im Computer modellieren, sodass wir vorhersagen können, mit welchen ‚Umbaumaßnahmen‘ eine Zelle auf Stress reagiert, zum Beispiel auf extreme Temperaturen oder auf eine Virusinfektion.« Ein zweites Ziel ist, mithilfe der erforschten Auf- und Abbauprozesse neuartige, synthetische Organellen zu schaffen, die neue Proteine, möglicherweise sogar Medikamente produzieren. »Und schließlich hoffen wir, dass SCALE dazu beiträgt, Krankheiten zu verstehen – und letztlich zu heilen –, die mit der krankhaften Veränderung der subzellulären Architektur zu tun haben.« Vermutet wird dies bei neurodegenerativen Erkrankungen wie etwa Parkinson und Alzheimer, bei denen sich im Zellplasma nicht lösliche Strukturen bilden, die dann komplett die Zelle verstopfen.
Wenn Zellen im Umbau sind
Solche Strukturen, sognannte Plaques aus dem Protein-Baustein Amyloid-Beta (Aβ), lagern sich etwa im Gehirn von Alzheimer-Patienten an. Damit beschäftigt sich das Projekt »Subzelluläre Architektur des Gefäßsystems in Gesundheit und Krankheit« der Chemikerin Prof. Dr. Nina Morgner und der Molekularbiologin Prof. Dr. Jasmin Hefendehl. Morgner untersucht mithilfe der Analyseverfahren »Ionen-Mobilitäts-Spektrometrie« und »Massen-Spektrometrie«, wie sich unterschiedlich lange Aβ-Bausteine zusammen lagern und schließlich Plaques bilden. Von der Länge der Aβ-Moleküle hängt insbesondere ab, wie toxisch diese wirken. »Speziell Aβ42, bei dem 42 Grund-Bausteine aneinanderhängen, wirkt sich besonders schädlich aus. Aβ40 schätzen wir als deutlich harmloser ein«, erläutert Morgner. Im Einzelnen zu verstehen, wie sich die Protein-Bausteine zu Aβ-Plaques verbinden, eröffne zudem die Möglichkeit, in diesen Vorgang einzugreifen. Das wiederum würde die Suche nach einem Wirkstoff für ein Alzheimer-Medikament entscheidend voranbringen.
An einer etwas anderen Stelle setzt Hefendehls Forschung zur toxischen Wirkung von Aβ an: Sie bringt verschieden lange Aβ-Ketten in Kontakt mit eben jener Art von Zellen, aus der die im Gehirn verlaufenden Blutgefäße bestehen. Gemeinsam mit Achilleas Frankgakis beobachtet sie, welche krankhaften Veränderungen in den Zellen auftreten. »Bei über 80 Prozent der Alzheimer-Patienten zeigt uns das Elektronenmikroskop Blutgefäßstörungen«, sagt Hefendehl. Sie vermutet, dass bei diesen Patienten die sogenannte Blut-Hirn-Schranke defekt ist. Das Gehirn wäre in diesem Fall der toxischen Wirkung der entzündungsauslösenden Proteine im Blut schutzlos ausgeliefert.
Die Experimente im Rahmen von SCALE sind erst durch jüngste technologische Fortschritte möglich geworden: »Unser Cluster profitiert entscheidend davon, dass die Kryo-Elektronenmikroskopie, also die Elektronenmikroskopie bei tiefen Temperaturen wie fast minus 200 Grad Celsius, in den vergangenen zehn Jahren riesige Fortschritte gemacht hat«, erläutert Müller-McNicoll. Mit diesem Verfahren ist es mittlerweile möglich, die Strukturen in Zellen und Zellmembranen mit atomarer Auflösung zu betrachten –, und zwar in ihrer nativen Umgebung: Die subzellulären Strukturen sind zwar tiefgefroren, aber die Forschenden beobachten die Zellen, ohne dass sie aufgereinigt, isoliert oder anderweitig vorbehandelt worden sind.
Die drei grundlegenden Themen des SCALE-Clusters erfordern einen interdisziplinären Zugang: So kommen die leitenden Wissenschaftler*innen aus den Forschungsbereichen Physik und Biologie sowie Chemie, Biochemie und Pharmazie. »Wir sind sehr froh, dass auch die Max-Planck-Institute für Biophysik und für Hirnforschung sowie das Frankfurt Institute of Advanced Studies bei SCALE mitarbeiten«, kommentiert Müller-McNicoll. »An unserem Projekt ist somit ein großer Teil der auf dem Riedberg ansässigen Institute beteiligt.«
(she)

Nachgefragt bei...
Michaela Müller-McNicoll
Welches Problem wollen Sie mit SCALE gern besser verstehen?
SCALE befasst sich mit subzellulärer Architektur, d.h. alle größeren Strukturen in einer Zelle, die entweder raumgebend sind oder eine spezifische Funktion ausüben. Dazu gehören Organellen wie das Mitochondrium, Kontakte zwischen Zellen, z.B. Synapsen oder Desmosomen, Zellkernporen und membranlose Kompartimente im Zellkern wie Chromatin und Nuclear Speckles. In SCALE wollen wir auf molekularer Ebene verstehen, wie diese subzellulären Strukturen entstehen, wie sie organisiert sind, wie sie abgebaut werden, welche Funktionen sie ausüben oder sich an verändernde Bedingungen anpassen.
Was ist Ihnen daran persönlich wichtig?
Ich untersuche die Rolle von verschiedenen Strukturen im Zellkern auch bezeichnet als nukleäre Architektur bei der Prozessierung von mRNA. Diese Strukturen entstehen oder verändern sich, wenn Zellen gestresst oder krank sind und ich möchte verstehen, welche Auswirkungen das auf die Expression von Genen hat.
Was ist ein Etappenziel?
Wir wollen Methoden und Technologien entwickeln, um Aktivitäten innerhalb von Zellen mit hoher räumlicher und zeitlicher Auflösung beobachten, lokal zu manipulieren und einer bestimmten Funktion zuzuordnen.
Was ist die größte Hürde?
Die Technologien sind sehr aufwendig und teuer und sind nur innerhalb von großen Konsortien finanzierbar.
Gibt es eine Erkenntnis, die Sie besonders geprägt hat und die Sie in dieses Projekt quasi als „Rüstzeug“ mitnehmen?
Interdisziplinäre Kollaborationen und die Nutzung von künstlicher Intelligenz sind essentiell, um die immer komplexeren Zusammenhänge in Zellen zu untersuchen und zu verstehen.
Prof. Dr. Michaela Müller McNicoll leitet die RNA-Biologie am Institut für Molekulare Biowissenschaften und ist gemeinsam mit Prof. Dr. Achilleas Frangakis Sprecher des Forschungsprojekts „SCALE“.

Beobachten, untersuchen, mikroskopieren – auch angehende Biologielehrer*innen sind in ihrem Studium auf Unterrichtsräume angewiesen. Was also tun, wenn pandemiebedingt „drinnen“ studieren nicht geht? Dann hilft „digital“ – oder eben „draußen“.
Schüler für Pflanzen begeistern – wie geht das? Unterricht zu planen, der alters- und interessengerecht ist, lernen angehende Biologielehrer*innen in fachdidaktischen Seminaren. Wie Schüler*innen biologisches Wissen sammeln, wie sie Versuche durchführen können, wie sie mit Mikroskopen umgehen, dies erproben, erfahren, konzipieren und reflektieren die Studierenden vorab in den Seminaren. In der Regel vor Ort, in den Räumen der Universität. In der Pandemie zeigte sich nun, dass sich Elemente eines fachdidaktischen Seminars auch nach Hause transportieren lassen. Einen Skelettmuskel nachbauen? Wärmeisolierung testen? Geht – mit etwas Fantasie und einigen Alltagsmaterialien wie Pfeifenreinigern und Federn oder Tierhaaren, die ein Gefäß mit warmem Wasser umgeben, lässt sich biologisches Wissen am Modell auf anschauliche Weise vermitteln – und anschließend per Foto und Video mit anderen teilen, um dann besprochen zu werden.
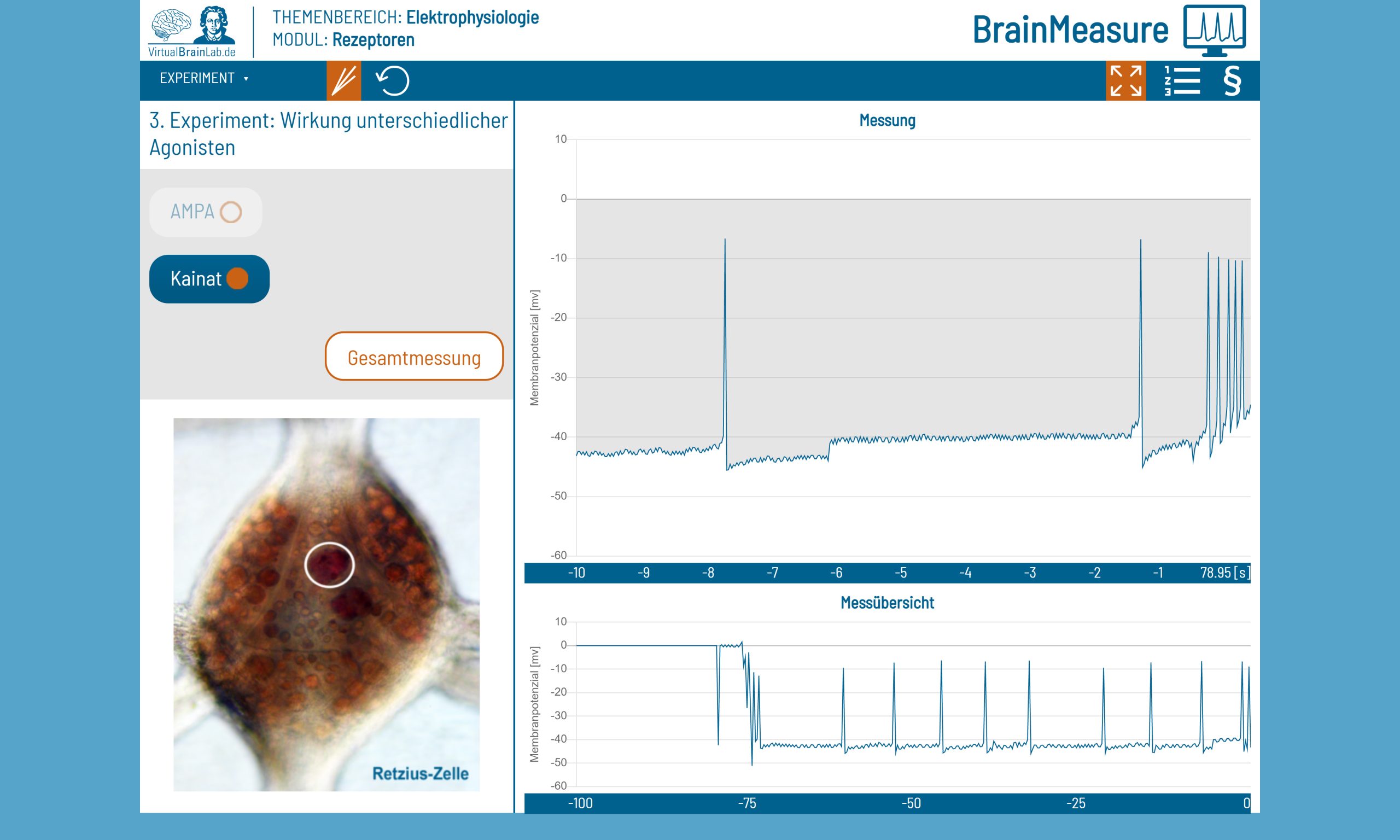
Wenn der Modellbau an Grenzen stößt, helfen digitale Versuchsbeschreibungen, Videodokumentationen und Online-Materialien weiter. So etwa wurde der Lehrstoff des Praktikums „Humanbiologie und Anthropologie“ durch das Online-Schülerlabor zum Thema Neurowissenschaften namens „VirtualBrainLab.de“ vermittelt. Das VirtualBrainLab wurde Ende 2020 von der Abteilung für Didaktik der Biowissenschaften und Zootierbiologie der Goethe-Universität für den Einsatz in Schulen entwickelt und dann an universitäre Lehrveranstaltungen angepasst. Originale Daten boten so die Grundlage für virtuelle elektrophysiologische Experimente und Reaktionen von Nervenzellen, die live verfolgt werden konnten. Beim Einsatz eines virtuellen Mikroskops konnten die Studierenden außerdem gesunde und pathologische Blutzellen vergleichen.
Bis zum Jahresende wurde das VirtualBrainLab übrigens von 563 Lehrkräften und 1070 Schüler*innen aus dem gesamten Bundesgebiet genutzt. Und es wurde prämiert: Vom Institut für Bildung und Medien der Gesellschaft für Pädagogik, Information und Medien e.V. erhielt es das Comenius EduMedia-Siegel in der Kategorie „didaktische digitale Medienprodukte“ für die Schul- und Hochschulbildung.

Pflanzen live, ergänzend zu virtuellen Lehrinhalten, bot für die Studierenden darüber hinaus das „Pet-Plant-Project“. Es sollte denjenigen, die eher zu Tieren einen Bezug haben, nicht nur ihre private Hauspflanze, sondern die Botanik im Allgemeinen näherbringen. Von der Aussaat bis zur Fruchtreife konnten Lehramtsstudierende ihre „Hauspflanze“ ein Semester lang begleiten und „kennenlernen“ – ein Lernmodell, welche einige Lehramtskandidat*innen modifiziert in den Schulalltag übertragen wollen.
Unsicherheit hat ihren Reiz
Biologieunterricht simulieren – das geht auch draußen im Freiland, auf dem Wildbienenlehrpfad, der Wildblumenwiese oder dem unieigenen Schulteich oder am fußläufig zu erreichenden Urselbach. Dort sollte dann von den Studierenden gekechert und bestimmt werden. Doch was im Urselbach an Lebendigem zu Tage gefördert wurde, stand nicht immer auf dem Lehrplan und konnte deshalb auch nicht immer umgehend identifiziert werden. „Das verunsichert manche Studierende und kann auch Dozenten ins Schwitzen bringen“, erklärt Biologiedidaktiker Prof. Dr. Volker Wenzel. „Wir haben aber gemerkt, dass diese Unsicherheit auch kreativ macht.“ Wer sich auf das Freiland einlasse, erweitere schneller sein Wissen zu Tier- und Pflanzenarten. Bestimmungs-Apps wie iNaturalist oder Flora Incognita können hier Lehrende wie Lernende unterstützen – „zumal wir uns in der Pandemie noch viel tiefer in die Möglichkeiten der Apps eingearbeitet haben und zum Beispiel eigene Projekte mit Studierenden anlegen können“, so Wenzel. „Wir sollten uns viel öfter den Primärerfahrungen mit Lebewesen aussetzen.“
Ein Highlight des Biologiestudiums für angehende Lehrkräfte ist die mehrtägige Exkursion, die etwa nach Helgoland oder ins Altmühltal führen kann. Das Ziel: Artenkenntnis erweitern, erlebnispädagogische Konzepte kennenlernen und schließlich Studienfahrten selbst planen lernen. 2020 schrumpften die gewohnten Reisestrecken auf einen Ausflug zum Campus Riedberg zusammen – wo das iNaturalist Biomonitoring-Projekt allerdings Geschichte schrieb: Innerhalb weniger Wochen wurden 948 unterschiedliche Pflanzen-, Tier- und Pilzarten auf dem Campus Riedberg identifiziert und kartographiert. Im folgenden Jahr waren bereits Tagesexkursionen möglich – zur Fossilienlagerstätte Welterbe Grube Messel etwa, zum Naturpark im Taunus und in den Tiergarten Weilburg. 2022 ist für Prof. Wenzel wieder Helgoland das Ziel. „Wir freuen uns wieder sehr auf ein gemeinsames Lernen“, sagt Wenzel. Den Reiz der Freilandbiologie und die Erfahrung, im unsicheren Terrain Überraschendes lernen zu können – „die nehmen wir mit“.

Unser Forschungsprofil: Raum für gute Antworten
Mit sechs Profilbereichen will die Goethe-Universität ihre Kompetenzen stärker bündeln.

