Hightech und Künstliche Intelligenz bringen Licht in den Nanokosmos
von Andreas Lorenz-Meyer
Der Chemiker Mike Heilemann will Prozesse in menschlichen Zellen besser verstehen, um die biomedizinische Forschung voranzubringen. Dabei arbeitet er mit superauflösender Mikroskopie, die das Unsichtbare sichtbar macht.
Im Jahr 1873 beschrieb der Jenaer Physiker Ernst Abbe folgendes Phänomen: Liegen zwei Strukturen enger beieinander als etwa die halbe Wellenlänge des zur Beobachtung eingesetzten Lichts, sind sie im Mikroskop nicht mehr räumlich voneinander getrennt abbildbar. Diese optische Auflösung liegt für sichtbares Licht im Bereich von 200 bis 300 Nanometern und sorgt dafür, dass näher zusammenliegende Strukturen zu einem einzigen Fleck verschwimmen. Das ist für die Zellbiologie schlecht, weil es eng zugeht in menschlichen Zellen. Ein fünf Nanometer kleines Protein zum Beispiel trennen von seinen Nachbarproteinen viel weniger als diese 200 Nanometer. Dicht gepackte Proteine in Zellen sind also mit herkömmlichen Technologien optisch nicht aufzulösen. Zum Glück aber lässt sich die Abbe’sche Beugungs- oder Auflösungsgrenze heute umgehen, mit trickreichen lichtmikroskopischen Verfahren, die unter dem Begriff superauflösende Mikroskopie zusammengefasst sind. Mike Heilemann vom Institut für Physikalische und Theoretische Chemie forscht genau in diesem Bereich. Er macht Schritt für Schritt immer mehr winzige Objekte und sogar räumliche Anordnungen im Nanokosmos Zelle sichtbar.
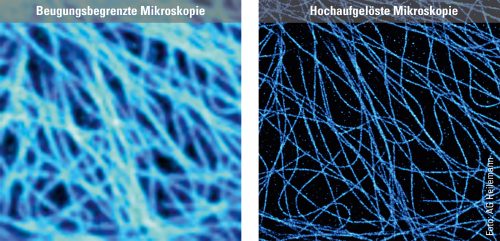
Um zu demonstrieren, wozu superauflösende Mikroskopie fähig ist, stellt der Chemiker in seinem Büro auf dem Computerbildschirm zwei Aufnahmen nebeneinander. Zu sehen ist jeweils das Strukturprotein Mikrotubulin, das in den Zellen so etwas wie Transportschienen bildet (Filamente), auf denen Stoffe von einem Ort zum anderen gelangen. Für die mikroskopischen Aufnahmen wurden die Filamente mit einem Fluoreszenzfarbstoff markiert, der im Laserlicht blau leuchtet. Das erste Bild zeigt die Grenzen klassischer Technologie auf. Alles erscheint reichlich verschwommen. Die Filamente, bläulich schimmernde Fäden, wirken so sehr aufgebläht, dass sie teils nicht auseinanderzuhalten sind. »Hier sehen wir die Beugung am Werk«, erklärt Heilemann. »Wenn Lichtwellen auf ein Hindernis treffen, ändern sie ihre Ausbreitungsrichtung. Das führt dazu, dass ein bis zwei Nanometer große Fluoreszenzfarbstoffe als viel größeres kreisförmiges Lichtmuster erscheinen, in einer Größe von 200 Nanometern. Genauso groß werden die dünnsten Tubulin-Filamente dargestellt, die eigentlich nur 25 Nanometer Durchmesser haben.« Anders schaut es beim zweitem Bild aus, das mit superauflösender Mikroskopie angefertigt wurde. Hier gibt es keinen »Aufbläheffekt« der Lichtsonden: Die Filamente erscheinen relativ scharf gestellt und können, weil sie dünn sind, im Bild voneinander getrennt wahrgenommen werden. So wird die räumliche Struktur sichtbar, das Wirrwarr der einzelnen, schlängelnd über- und untereinander laufenden Fäden.
Viele Aufnahmen hintereinander
Eine wichtige Technologie der superauflösenden Mikroskopie ist die Einzelmolekül-Lokalisierungsmikroskopie (single molecule localization microscopy, SMLM), ein spezielles fluorenszenzmikroskopisches Verfahren. Im Kern werden bei der Fluoreszenzmikroskopie Farbstoffmoleküle verwandt, die von Licht angeregt selbst Licht mit veränderter Wellenlänge abgeben (Fluoreszenz). Man hängt den Farbstoff an ein Biomolekül und steuert beide zusammen, als Fluoreszenzsonde, in Richtung Zielmolekül in der Zelle, wo die Sonde andockt. Wird sie durch Laserlicht bestimmter Wellenlängen angestrahlt, schießt sie Lichtblitze ab, und so wird das Zielmolekül, etwa ein Protein, im Mikroskop sichtbar. Der Trick bei SMLM: Die Sonden geben die Lichtblitze nicht gleichzeitig ab, sondern hintereinander. Ein Zielmolekül nach dem anderen leuchtet auf. »Diese zeitliche Trennung erlaubt es, Bilder zu rekonstruieren, die eine nahezu molekulare Auflösung von wenigen Nanometern haben«, so Heilemann.
Einzelmolekül-Lokalisierungsmikroskopie hat jedoch einen großen Nachteil: Sie ist langsam. Heilemann erläutert: »Nehmen wir an, unser Zielprotein in einer einzelnen Zelle tritt in 100 000 Kopien auf. Um dieses abzubilden, müssen wir insgesamt 100 000 einzelne Punkte optisch voneinander trennen. Pro Bildaufnahme können nur wenige Moleküle gleichzeitig detektiert werden, sonst kommt es zu Überlappungen.« Die Aufnahme würde dann nur etwas undefinierbar Wolkenartiges zeigen und nicht die molekulare Struktur. Nur wenige Moleküle pro Bildaufnahme bedeuten aber, dass bei 100 000 Punkten viele einzelne Aufnahmen nötig sind – eine zeitraubende Angelegenheit.
Künstliche Intelligenz hilft
Heilemann ist es jedoch gelungen, das Verfahren erheblich zu beschleunigen. Als Grundtechnik setzt er einen SMLM-Ableger ein, das PAINT-Verfahren. Hier docken die Fluoreszenzsonden nur ganz kurz ans Zielmolekül an, geben ihr Lichtsignal ab und schwimmen dann wieder weg. Tempomacher sind neuronale Netzwerke (Deep Learning), die als »digitale Erweiterung« ans Mikroskop gebaut wurden. Diese Künstliche Intelligenz lässt sich so trainieren, dass sie Moleküle erkennt und deren Position bestimmt, selbst wenn der Abstand zwischen den Molekülen viel kleiner ist als die Auflösungsgrenze. »So können wir eine viel größere Anzahl an Molekülen pro Bildaufnahme verarbeiten«, erklärt Heilemann. Die Bildgebung wird 10 bis 20 Mal so schnell, und es sind nur noch wenige einzelne Aufnahmen für die komplette Struktur notwendig.
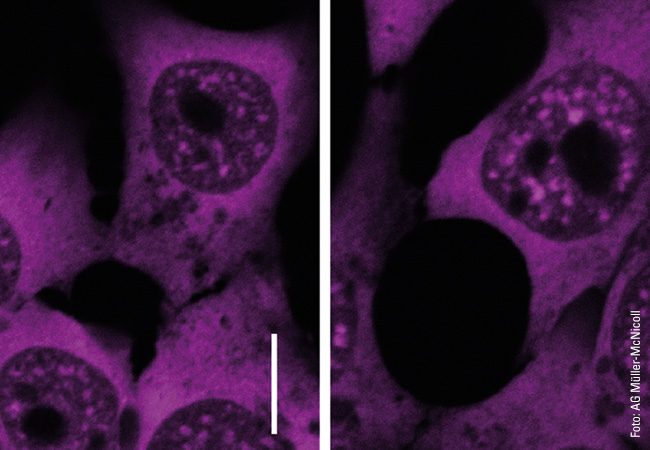
Noch viel wichtiger als die Zeitersparnis ist für Heilemann und sein Team aber der wissenschaftliche Erkenntnisgewinn, den die neuronalen Netze bringen. Denn erst durch die Tempoerhöhung lassen sich Dynamiken visualisieren, die Bewegungen und Veränderungen in der lebenden Zelle. Dies veranschaulicht ein hochaufgelöstes Video einer lebenden Zelle, das Heilemann auf seinem Rechner zeigt: Zu sehen ist ein Organell im Zytoplasma, das Endoplasmatische Reticulum. Es besteht aus Membranen, die viele Röhren und Taschen bilden. Im Video sind die Röhren als dünne, weiße Linien zu erkennen und pausenlos in Bewegung. Sie trennen sich voneinander und verbinden sich neu. In jeder Sekunde verändert das Gebilde seine Form. So sieht es aus, wenn sich die Röhren des Organells reorganisieren. Im Leben einer Zelle läuft dieser Prozess permanent ab. Er hängt mit den Aufgaben des Endoplasmatischen Reticulums zusammen, das für Teilschritte der Proteinsynthese, aber auch für den Proteinabbau verantwortlich ist. »Vieles, was da passiert, verstehen wir noch nicht, da wir mit der Bildgebung erst seit Kurzem in diese Dimension vorgedrungen sind. Derzeit schaffen wir 30 bis 40 Nanometer Auflösung – ein sehr guter Wert für die Strukturbildgebung in lebenden Zellen. Entscheidend ist dabei, dass wir erneuerbare Fluoreszenzsonden verwenden, sodass wir diese Dynamik für eine lange Zeit in lebenden Zellen beobachten können.«
Unterschiedliche Farben kommen nachher dazu
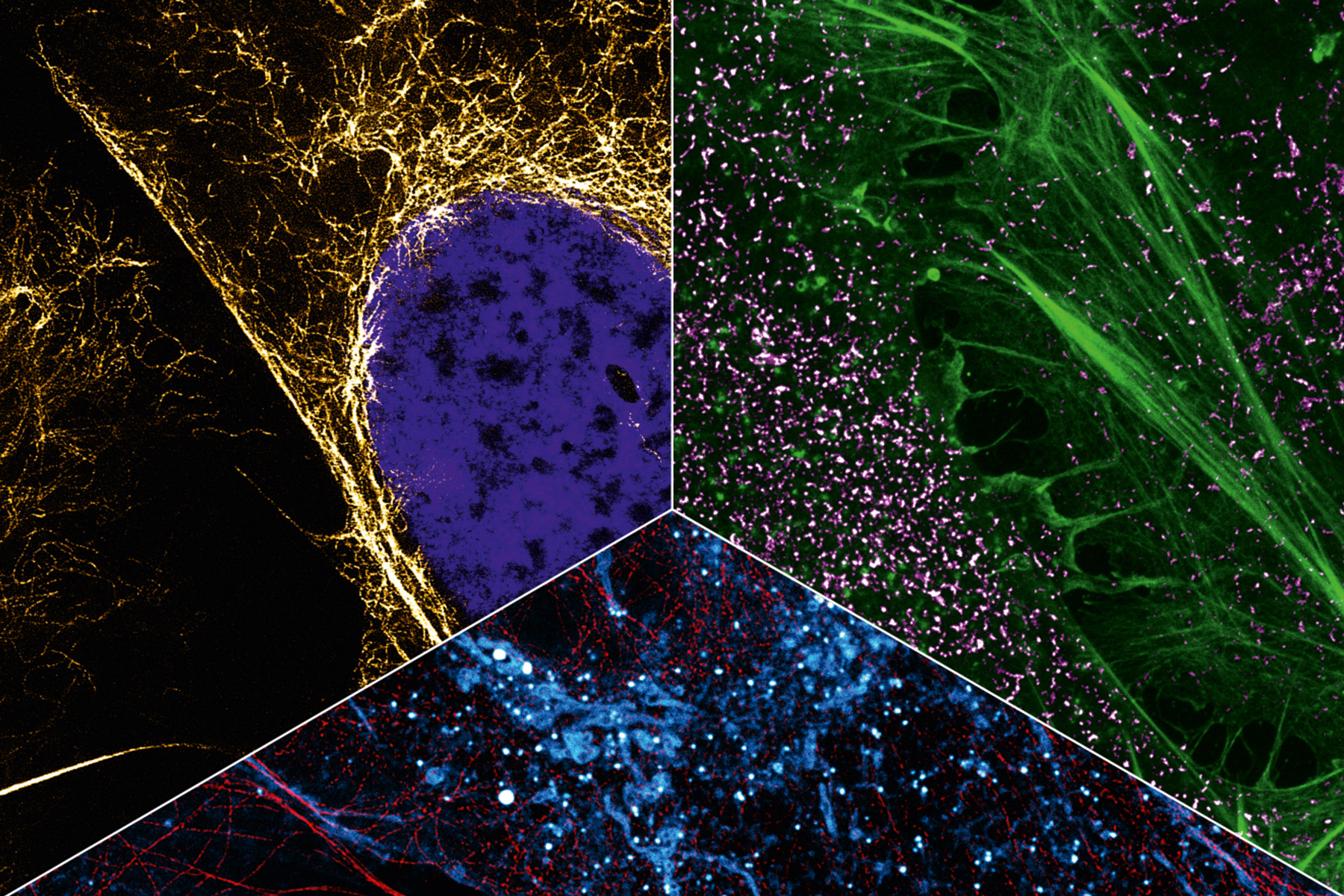
Bei Heilemanns Arbeit geht es neben dem Einfangen der Dynamik auch darum, zelluläre Objekte innerhalb einer Aufnahme unterscheiden zu können – und dafür braucht es Farben, möglichst viele Farben. Heilemann: »In jeder Zelle gibt es Tausende verschiedener Proteine mit unterschiedlichen Funktionen. Es reicht nicht, ein oder zwei davon zu betrachten, denn wir wollen die ›molekulare Soziologie‹ verstehen, das Zusammenspiel der einzelnen Proteine und Proteinkomplexe. Diese sind, wie wir Menschen, letztlich nur das Resultat ihrer Umgebung. Und diese Umgebung müssen wir als Ganzes abbilden, um die strukturelle Organisation der Zelle zu entschlüsseln.«
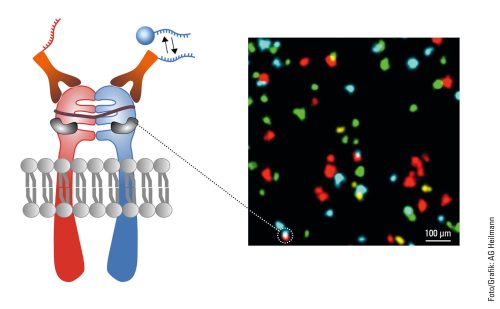
Hierzu setzen der Forscher und sein Team auch wieder das PAINT-Verfahren ein, welches Zielmolekül und Lichtsonde ja nur für eine kurze Zeit zusammenbringt – anders als in der klassischen Fluoreszenzmikroskopie, wo Lichtsonde und Zielmolekül fest miteinander verknüpft sind. Als Sonden dienen kurze DNA-Stränge. Weil diese PAINT-typisch nur kurz binden und dann wieder wegdiffundieren, blockieren sie das Zielmolekül nicht. Auf diese Weise lassen sich viele Proteine nacheinander in der gleichen Zelle visualisieren, das Verfahren nennt sich Multiplexing. »Dadurch können wir nacheinander eine größere Zahl an Proteinen visualisieren als bei herkömmlicher Fluoreszenzmikroskopie.« Die Proteine werden dann nach der eigentlichen Visualisierung noch mit Farben versehen – jeder Proteintyp bekommt dabei einen eigenen Anstrich.
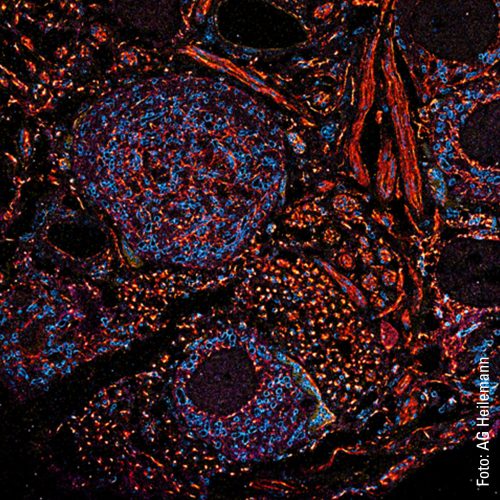
Eine Multiplexing-Aufnahme zeigt es: Zu sehen sind vor schwarzem Hintergrund viele kleine Punkte in den Farben Grün, Gelb, Rot und Blau. Wie ein bunter Nachthimmel voll blinkender Sterne. Die »Sterne« sind in diesem Fall spezielle Proteine in der Zellmembran, die Fibroblasten-Wachstumsfaktor-Rezeptoren (fibroblast growth factor receptors, FGFR). Von denen gibt es vier verschiedene. Die roten Punkte sind FGFR1, die gelben FGFR2, die grünen FGFR3 und die roten FGFR4. Man sieht, dass sich manche Proteine in räumlicher Nähe befinden. Es gibt ein rot-blaues Proteingespann, genauso ein rot-grünes und ein blau-gelbes. Auch solche Interaktionen sind mit Multiplexing visualisierbar. Und wenn mal komplexere Strukturen mit mehr als vier unterschiedlichen Proteintypen sichtbar gemacht werden sollen? Kein Problem, so Heilemann: »Wir können diese Technik erweitern und noch viel mehr Proteine darstellen.«
Die Lichtblitze zählen
Als Nächstes versuchen Heilemann und sein Team, versteckte Informationen mithilfe physikalischer »Tricks« aus den Aufnahmen herauszufiltern. Denn ihnen ist etwas aufgefallen: Eine Fluoreszenzsonde an einem Zielmolekül gibt nicht nur einen Lichtblitz ab. Es sind meist drei, vier oder sogar fünf Signale. »Die Frequenz verrät uns die Anzahl der Moleküle an dieser Position. Das heißt, wir werden da Objekte finden, die wir räumlich selbst mit hochauflösender Mikroskopie gar nicht auflösen können.« Ein weiterer wichtiger Schritt: die Untersuchung von molekularen Prozessen auf der Nanoskala in sowohl gesunden als auch kranken Zellen. Was in einer gesunden Zelle passiert, ist ja etwas anders als der Prozess in einer kranken. Die Unterschiede sollen herausgearbeitet werden. Zudem ist geplant, die Wirkung von Substanzen auf diese Prozesse zu analysieren. Die Grundlagenforschung werde mithilfe superauflösender Mikroskopie große Fortschritte machen, hofft Heilemann. Das Verständnis der genauen Zusammensetzung von Proteinkomplexen und deren Dynamik stelle in Zukunft eine wichtige Basis für die Entwicklung zielgenauer Wirkstoffe gegen Krankheiten dar.

Zur Person
Mike Heilemann hat in Konstanz, Heidelberg und Montpellier von 1996 bis 2001 Chemie studiert und von 2002 bis 2005 im Fach Physik in Heidelberg und Bielefeld seine Promotion angefertigt. Es folgten Forschungsaufenthalte in Oxford, Bielefeld und Würzburg. Seit 2012 ist er Professor am Institut für Physikalische und Theoretische Chemie. Er ist Mitglied der Bunsengesellschaft für Physikalische Chemie, der European Light Microscopy Initiative, der Biophysical Society, der European Photophysical Association und der Society of Photo-Optical Instrumentation Engineers. Heilemann ist Principal Investigator der Clusterinitiative SCALE (https://scale-frankfurt.org) der Goethe-Universität. Der Forschungsverbund entwickelt neuartige Technologien, um die inneren Strukturen von Zellen abzubilden und ihr Verhalten vorherzusagen.
Der Autor
Andreas Lorenz-Meyer, Jahrgang 1974, wohnt in der Pfalz und arbeitet seit 13 Jahren als freischaffender Journalist mit Schwerpunkt Nachhaltigkeit, Klimakrise, erneuerbare Energien, Digitalisierung. Er veröffentlicht in Tageszeitungen, Fachzeitungen, Universitäts- und Jugendmagazinen.