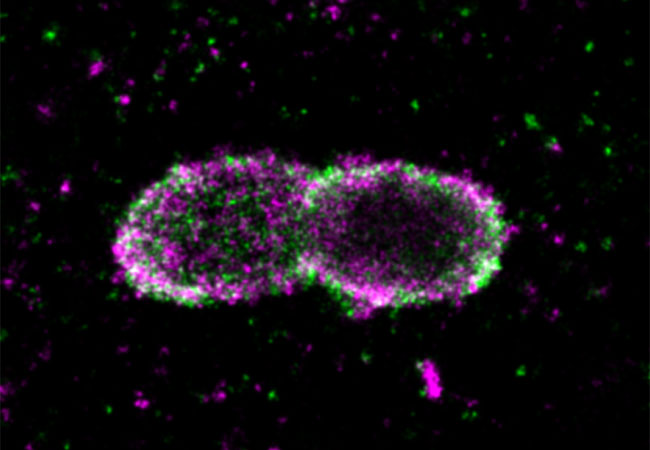
Bakterielle Infektionen stellen eine enorme globale Bedrohung dar, weltweit nimmt die Verbreitung von Bakterienstämmen zu, die gegen gängige Antibiotika resistent sind. Forscher hoffen, neuartige Therapien zu finden, indem sie die Wechselwirkung zwischen Erreger und Wirt im Detail verstehen lernen. Nun haben Wissenschaftler der Goethe-Universität am Beispiel der Salmonellen-Infektion einen entscheidenden Beitrag dazu geleistet.
Bakterien haben ausgeklügelte Mechanismen entwickelt, um in Wirtszellen zu überleben und sich zu vermehren. So verbergen sich Salmonellen innerhalb der Zelle normalerweise in membranumhüllten Körperchen, nur wenige brechen in das Zellinnere aus. Diese „Ausbrecher“ sind jedoch besonders gefährlich, weil sie sich rasend schnell vermehren und ausbreiten können. Die Zelle hat wirkungsvolle Abwehrmechanismen entwickelt, um eine solche Invasion zu stoppen. Ein interdisziplinäres Team um Prof. Ivan Dikic vom Institut für Biochemie II und Prof. Mike Heilemann vom Institut für Physikalische und Theoretische Chemie der Goethe-Universität hat jetzt einen dieser Mechanismen aufgeklärt.
Protein-Ketten geben Signale für Entzündungsantwort weiter
Die Zelle reagiert schnell: Sie markiert die ausgebrochenen Bakterien mit einem kleinen Protein, dem Ubiquitin, das in viele zelluläre Prozesse regulierend eingreift. Diese Markierung mit Ubiquitin erfolgt in langen, unterschiedlich verzweigten Ketten. Daraus ergibt sich ein regelrechter Geheimcode, der noch längst nicht vollständig entschlüsselt ist. Die Ubiquitin-Ketten geben – ähnlich wie Funkmasten – spezifische Signale weiter.
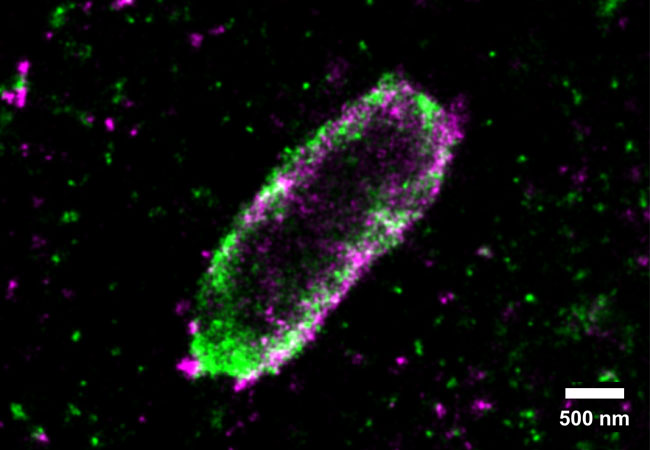
Mit hochauflösender Mikroskopie ist es dem Frankfurter Team nun erstmals gelungen, die verschiedenartigen Ubiquitin-Markierungen rund um Salmonellen sichtbar zu machen und deren molekulare Organisation detailliert zu untersuchen. Sie stellten fest, dass bei einer bakteriellen Invasion ein ganz bestimmter Kettentypus, die sogenannten linearen Ubiquitin-Ketten, eine wichtige Rolle spielen: Sie leiten den Abbau der Bakterien ein und lösen zusätzlich eine Entzündungsantwort aus. Dadurch werden die verbleibenden Bakterien in Schach gehalten. Gleichzeitig identifizierten die Forscher das Enzym Otulin als wichtigen Regulator. Otulin vermag die Weiterleitung von Signalen zu stoppen, was für eine exakte Kontrolle der Entzündungsantwort essentiell ist. Damit wird vermieden, dass eine überschießende Entzündung ähnlich viel oder sogar mehr Schaden anrichtet als der bakterielle Erreger selbst.
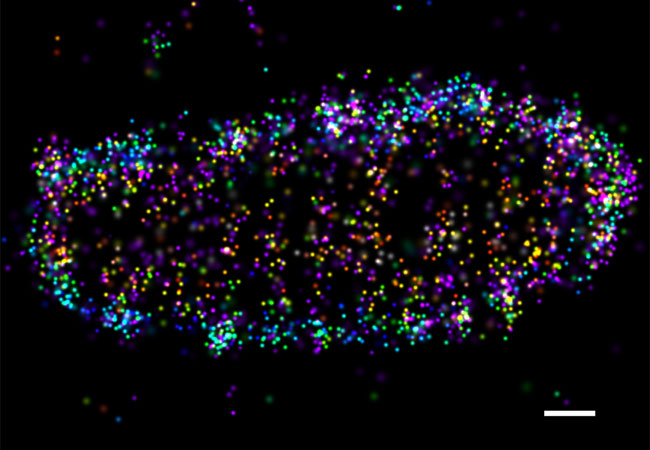
Diese neuen Erkenntnisse öffnen nun den Weg für zahlreiche weiterführende Projekte und könnten unter anderem eines Tages den Grundstein für neue therapeutische Ansätze liefern. Erst kürzlich erhielt Ivan Dikic einen der renommierten ERC Advanced Grants in Höhe von 2,5 Millionen Euro, um die Rolle der Ubiquitinierung bei bakteriellen Infektionen weiter zu erforschen.
Die Signalweiterleitung durch Ubiquitin spielt aber nicht nur bei der Abwehr von Infektionen eine bedeutende Rolle, sondern auch bei immunologischen und neurodegenerativen Erkrankungen und bei Krebs. Bislang ist jedoch nur ansatzweise bekannt, wie aus kleinen Fehlern in diesem System schwere Krankheiten entstehen und wie man gezielt therapeutisch eingreifen kann.
Verschiedene Förderungen ermöglichten das interdisziplinäre Forschungsprojekt. Die Frankfurter Forschungen wurden unter anderem durch Unterstützung des LOEWE-Schwerpunktes Ubiquitin-Netzwerke, des Sonderforschungsbereichs 1177 zur selektiven Autophagie und des Exzellenzclusters Makromolekulare Komplexe ermöglicht.
Die Ergebnisse werden in der aktuellen Ausgabe von Nature Microbiology publiziert, parallel zu den neuesten Erkenntnissen einer befreundeten Arbeitsgruppe aus Cambridge (England), die weitere Details zur linearen Ubiquitinierung von Bakterien enthüllt.
[dt_call_to_action content_size=“small“ background=“fancy“ line=“true“ style=“1″ animation=“fadeIn“]
Publikation: van Wijk SJ, Fricke F, Herhaus L, Gupta J, Hötte K, Pampaloni F, Grumati P, Kaulich M, Sou Y, Komatsu M, Greten F, Fulda S, Heilemann M, Dikic I. Linear ubiquitination of cytosolic Salmonella Typhimurium activates NF-κB and restricts bacterial proliferation. Nature Microbiology 2017, doi 10.1038/nmicrobiol.2017.66.
[/dt_call_to_action]
Quelle: Pressemitteilung vom 8. Mai 2017