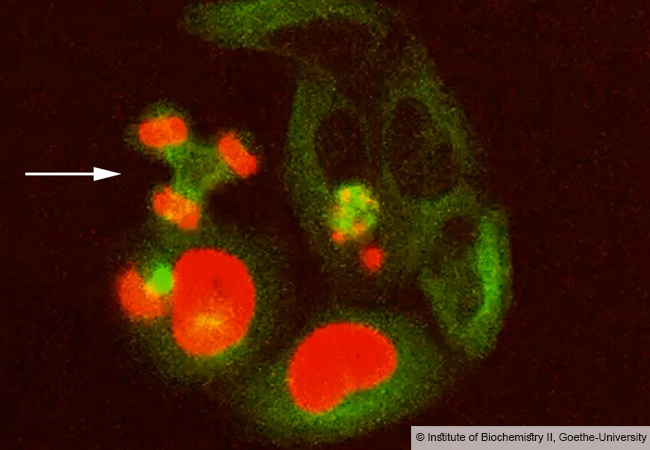
Mikroorganismen bauen Naturstoffe oft wie am Fließband zusammen. Dabei spielen bestimmte Enzyme, die nicht-ribosomalen Peptid Synthetasen (NRPS), eine Schlüsselrolle. Biotechnologen der Goethe-Universität ist es jetzt gelungen, diese Enzyme so zu verändern, dass ganz neue Naturstoffe oder auch Bibliotheken von Naturstoffen entstehen.
Viele wichtige Naturstoffe wie Antibiotika, Immunsuppressiva oder Mittel gegen Krebs stammen aus dem Stoffwechsel von Mikroorganismen. Dabei handelt es sich meistens um kleine Eiweißmoleküle (Peptide), die häufig so komplex sind, dass sie durch chemische Synthese im Labor nicht, oder nur mit großem Aufwand hergestellt werden könnten. In der Zelle entstehen sie mit Hilfe der NRPS-Enzyme wie am Fließband in einer modernen Autofabrik: an jeder Station werden dem Grundgerüst weitere Teile zugefügt, bis am Ende das fertige Auto aus der Fabrik rollt. Im Fall der NRPS findet an jeder Station (genannt Modul) der Einbau und die Prozessierung einer bestimmten Aminosäure statt, so dass am Ende ketten-, ringförmige oder anders modifizierte Peptide entstehen, die auch ungewöhnliche Aminosäuren tragen können.
Obwohl die grundlegenden Prinzipien der NRPS schon lange bekannt sind, war es bisher kaum möglich, diese Enzyme einfach und effizient zu modifizieren oder die Enzyme komplett neu zusammenzusetzen, so dass die Mikroorganismen neuartige Peptide in guten Ausbeuten produzieren. Die Arbeitsgruppe von Prof. Helge Bode, Professur für Molekulare Biotechnologie, hat nun ein von ihr 2018 publiziertes Verfahren weiterentwickelt, mit dem dies noch einfacher möglich ist.
„Wir nutzen Fragmente natürlicher NRPS-Systeme aus Bakterien als Bausteine, die wir über von uns identifizierte Schnittstellen neu zusammenfügen“, erläutern Andreas Tietze und Janik Kranz den Forschungsansatz, den beide mitentwickelt haben. Die Ausbeuten sind dabei vergleichbar mit der natürlichen Produktion dieser Naturstoffe. Mit der neuen Methode können nun auch Peptidbibliotheken sehr einfach erzeugt werden, was vorher nicht möglich war.
Die Methode ist inzwischen so gut ausgearbeitet, dass sie von Anfängern bereits nach kurzer Einarbeitung zur Herstellung neuer Peptide genutzt werden kann. Allerdings war es bis dahin ein weiter Weg. „Nachdem die ersten Versuche meines damaligen Doktoranden Kenan sehr vielversprechend waren, haben wir lange mit einem Großteil meiner Gruppe an dem Projekt gearbeitet, bis wir uns sicher waren, dass es die Erwartungen an eine robuste und gut reproduzierbare Methode erfüllt“, erklärt Bode. „Dank des LOEWE Schwerpunkts MegaSyn und des LOEWE Zentrums Translationale Biodiversitätsgenomik (TBG) hatten wir die notwenige personelle und finanzielle Unterstützung und konnten uns ganz auf das Projekt konzentrieren“.
Nun geht es darum, die ersten klinisch relevanten Wirkstoffe mit dieser Methode zu modifizieren und biotechnologisch herzustellen. Die Voraussetzungen dafür sind gut, hat Bode doch kürzlich einen der renommierten ERC Advanced Grants des Europäischen Forschungsrats eingeworben, um die Methoden in den nächsten fünf Jahren weiter zu optimieren.
Publikation: Kenan A. J. Bozhüyük, Annabell Linck, Andreas Tietze, Janik Kranz, Frank Wesche, Sarah Nowak, Florian Fleischhacker, Yan-Ni Shi, Peter Grün, Helge B. Bode: Modification and de novo design of non-ribosomal peptide synthetases (NRPS) using specific assembly points within condensation domains, Nature Chemistry, https://www.nature.com/articles/s41557-019-0276-z; DOI: https://doi.org/10.1038/s41557-019-0276-z