Mikrobiologen der Goethe-Universität haben zusammen mit Forschern aus Marburg und Basel die Struktur eines Enzyms aufgeklärt, das aus molekularem Wasserstoff (H2) und Kohlendioxid (CO2) Ameisensäure bildet. Das Enzym des Bakteriums Thermoanaerobacter kivui war vor einigen Jahren von Mikrobiologen der Goethe-Universität entdeckt worden. Kürzlich hatten die Wissenschaftler sein Potenzial zur Herstellung eines flüssigen Wasserstoffspeichers vorgestellt. Die jetzt erstmals atomgenau beschriebene fadenförmige Struktur des Enzyms wirkt wie ein Nanodraht und ist offenbar für die extrem effiziente Umwandlung der beiden Gase verantwortlich (Nature, DOI 10.1038/s41586-022-04971-z).

2013 entdeckten Mikrobiologen um Prof. Volker Müller von der Goethe-Universität ein außergewöhnliches Enzym in einem wärmeliebenden (thermophilen) Bakterium: die wasserstoffabhängige CO2-Reduktase HDCR. Sie stellt aus gasförmigem Wasserstoff (H2) und Kohlendioxid (CO2) Ameisensäure (Formiat) her, wobei der Wasserstoff Elektronen an das Kohlendioxid überträgt. Damit ist die HDCR das erste bekannte Enzym, das direkt Wasserstoff nutzen kann. Alle bis dahin bekannten Enzyme, die Ameisensäure produzieren, nutzen dagegen einen Umweg: Sie erhalten die Elektronen von löslichen zellulären Elektronenüberträgern, die die Elektronen ihrerseits mit Hilfe weiterer Enzyme vom Wasserstoff empfangen.
Das Bakterium Thermoanaerobacter kivui lebtfern vom Sauerstoff zum Beispiel in der Tiefsee und nutzt CO2 und Wasserstoff zur Energiegewinnung. Die HDCR von Thermoanaerobacter kivui besteht aus 4 Protein-Modulen, einem wasserstoffspaltenden und einem ameisensäurebildenden Modul und zwei kleinen Eisen-Schwefel-haltigen Modulen. „Schon nach der Entdeckung war uns klar, dass es die beiden kleinen Untereinheiten sein müssen, die Elektronen vom einen zum anderen Modul leiten“, sagt Müller. 2016 beobachteten die Forscher, dass das Enzym lange Fäden (Filamente) bildet. Müller: „Wie wichtig diese Struktur war, konnten wir daran erkennen, dass die Fadenbildung die Enzymaktivität stark stimuliert.“
Jetzt haben die Forscher der Goethe-Universität zusammen mit der Gruppe von Dr. Jan Schuller, Philipps-Universität Marburg und LOEWE Zentrum für Synthetische Mikrobiologie, eine molekulare Nahaufnahme des Enzyms gemacht. Durch kryo-elektronenmikroskopische Analysen gelang Schullers Gruppe die Bestimmung der HDCR-Raumstruktur in atomgenauer Auflösung. Damit wurden die Details der langen Filamente sichtbar, die das Enzym unter den experimentellen Bedingungen im Labor (in vitro) bildet: Das Rückgrat der Filamente besteht aus den beiden kleinen Untereinheiten der HDCR, die so zu einer Art Nanodraht mit Tausenden von elektronen-leitenden Eisenatomen zusammengelagert sind. „Dies ist der bisher einzige enzymatisch dekorierte Nanodraht. Auf diesem Draht sitzen das Hydrogenase-Modul und das Formiat-Dehydrogenasemodul wie Pilzköpfe auf einer Leitung“, erklärt Schuller.
Helge Dietrich, Doktorand an der Goethe-Universität in der Gruppe von Volker Müller, testete eine genetische Modifikation der kleinen Module, die die Bildung der HDCR-Fäden verhinderte. Das Ergebnis: Die einzelnen Bausteine oder Monomere waren weit weniger aktiv als das Filament.
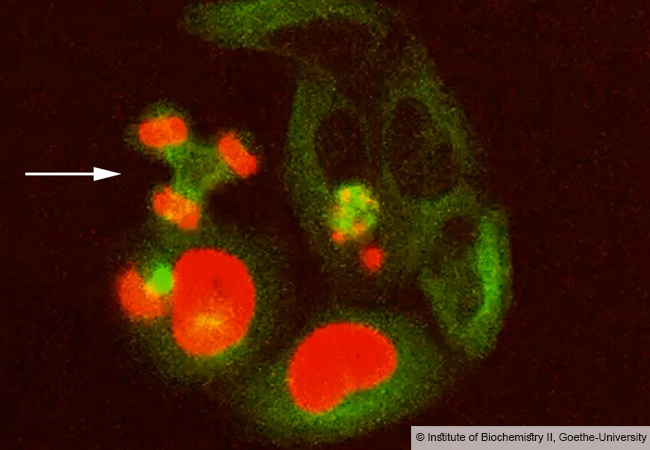
Auch in Bakterienzellen lagern sich Enzym-Monomere zu Fadenstrukturen zusammen. Diese Erkenntnis steuerten der Baseler Zellstrukturbiologe Prof. Ben Engel und sein Team durch kryo-elektronentomografische Untersuchungen bei. Mithilfe dieser Spitzentechnologie gelang den Forschern eine besondere Entdeckung: „Hunderte dieser Filamente sind umeinandergewunden und bilden übergeordnete ringförmige Strukturen“, erklärt Engel. Die Bündel sind offensichtlich in der inneren Membran der Bakterienzelle verankert und durchspannen fast die gesamte Breite der Zelle. Dr. Ricardo Righetto, Wissenschaftler in Ben Engels Team, analysierte, wie diese Strukturen aus HDCR-Filamenten in Bakterien aussehen: „Mithilfe der Kryo-Elektronentomografie konnten wir mit hoher Auflösung direkt in die Zellen hineinsehen. Wir waren überrascht, dass wir nicht nur die Bildung HDCR-Filamente bestätigen konnten, sondern auch, dass sie große Bündel bilden, die in der Membran verankert sind.“
Mit der Struktur wird deutlich, woran es liegt, dass HDCR um Größenordnungen effizienter als alle chemischen Katalysatoren und deutlich besser als alle bekannten Enzyme Ameisensäure als „flüssigen Wasserstoffspeicher“ aus Wasserstoff und CO2 herstellen kann (vgl. Hintergrundinformation). Volker Müller: „Die Wasserstoffkonzentrationen im Ökosystem dieser Bakterien sind gering, und darüber hinaus können die CO2– und H2 -Konzentrationen wechseln. Die Bildung und darüber hinaus die Bündelung der Filamente schaffen nicht nur eine deutliche Erhöhung der Konzentration dieser Enzyme in der Zelle. Die Tausenden von elektronen-leitenden Eisenatomen in diesem ‚Nanodraht‘ können auch die Elektronen aus der Wasserstoffoxidation zwischenspeichern, wenn gerade mal eine Wasserstoffblase an den Bakterien vorbeizieht.“
Durch die atomare Auflösung der Struktur sind die Rätsel der HDCR noch nicht alle gelöst, ist das Team überzeugt. Jan Schuller meint: „Wir wissen noch nicht, wie der Draht die Elektronen speichert, warum die enzymatische Aktivität durch die Filamentbildung so stark stimuliert wird und wie die Bündel in der Membran verankert sind. An diesen Forschungsfragen arbeiten wir.“ Doch die Zukunft der HDCR könnte sehr spannend werden, glaubt Volker Müller: „Vielleicht können wir einmal synthetische Nanodrähte herstellen, mit denen wir CO2 aus der Atmosphäre einfangen können. Auch die biologische Wasserstoffspeicherung ist jetzt einen Schritt näher gerückt.“
Publikation: Helge M. Dietrich, Ricardo D. Righetto, Anuj Kumar, Wojciech Wietrzynski, Raphael Trischler, Sandra K. Schuller, Jonathan Wagner, Fabian M. Schwarz, Benjamin D. Engel, Volker Müller & Jan M. Schuller. Membrane-anchored HDCR nanowires drive hydrogen-powered CO2 fixation. Nature (2022) https://doi.org/10.1038/s41586-022-04971-z