Struktur eines für die Reifung von tRNA essenziellen Enzyms gibt Einblicke in die Ursache neurodegenerativer Erkrankungen
Das Biomolekül Transfer-RNA (tRNA) spielt in allen Lebewesen eine entscheidende Rolle bei der Produktion von Eiweißen. Die tRNAs entstehen in mehreren Schritten aus Vorläufermolekülen. Katalysiert wird der Prozess unter anderem durch das Enzym tRNA Splicing Endonuclease (TSEN). Defekte in TSEN führen zu einer neurodegenerativen Erkrankung, der Pontozerebellären Hypoplasie, die mit schweren Behinderungen und einem frühen Tod einhergeht. Forscher der Goethe-Universität Frankfurt und der Johannes Gutenberg-Universität Mainz haben nun aus der Struktur von TSEN dessen Funktionsweise abgeleitet und damit einen möglichen Weg zur Wirkstoffsuche gegen die Pontozerebelläre Hypoplasie eröffnet.
Die Transfer-RNAs (tRNAs) gehören zu den häufigsten RNA-Arten in einer Zelle und sind bei allen bekannten Organismen unverzichtbar für die Herstellung von Eiweißen. Dabei haben sie eine entscheidende „Übersetzungs“-Funktion: Sie bestimmen, wie die Reihenfolge der Nukleinsäuren, in denen die genetische Information kodiert ist, in eine Folge von Aminosäuren umgeschrieben wird, aus denen Eiweiße aufgebaut sind.
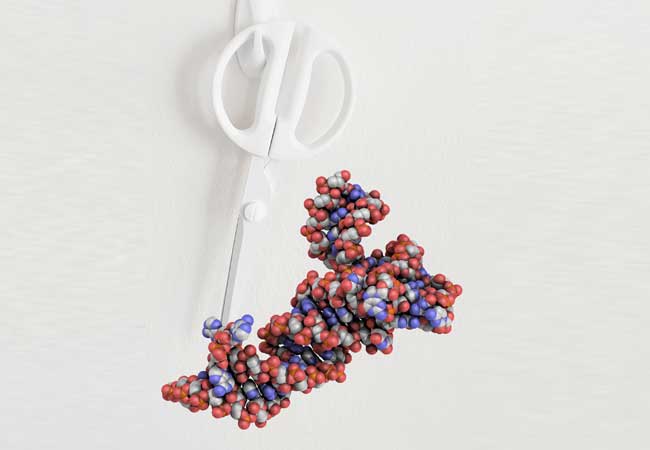
tRNAs entstehen aus Rohlingen (prä-tRNAs), die in mehreren Schritten zur fertigen tRNA mit komplexer dreidimensionaler Struktur umgebaut werden. Bei einigen tRNAs gehört dazu ein Schritt, bei dem ein bestimmter Abschnitt der prä-tRNA herausgeschnitten wird, ein sogenanntes Intron. Diese als Spleißen (engl. Splicing) bezeichnete Aufgabe übernimmt beim Menschen die tRNA Splicing Endonuclease TSEN.
Für den korrekten Umbau der tRNAs spielt außerdem das Enzym RNA-Kinase CLP1 eine Rolle, das mit TSEN eine direkte Verbindung eingeht. Können TSEN und CLP1 aufgrund genetischer Defekte nicht miteinander wechselwirken, scheinen auch tRNAs nicht mehr korrekt hergestellt werden zu können. Die Folgen zeigen sich häufig in der Ausbildung neurodegenerativer Erkrankungen. Eine davon ist die Pontozerebelläre Hypoplasie, die bereits in frühester Kindheit zu schweren Behinderungen und einem frühen Tod führt. Die sehr seltene fortschreitende Erkrankung äußert sich in einer Fehlentwicklung von Kleinhirn und einem Teil des Hirnstamms, der sogenannten Brücke (Pons).
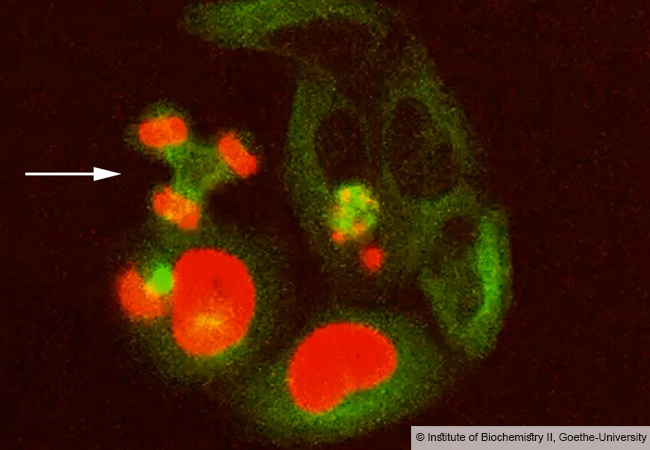
Obwohl die Aktivität von TSEN lebenswichtig ist, lag bislang weitgehend im Unklaren, wie das Enzym die prä-tRNA bindet und wie daraufhin das Intron aus der prä-tRNA herausgeschnitten wird. Das Fehlen einer dreidimensionalen Struktur des Enzyms machte es auch schwierig, die Veränderungen abzuschätzen, die bestimmte, krankmachende Mutationen auslösen. Nun ist es Forschern um Dr. Simon Trowitzsch vom Institut für Biochemie der Goethe-Universität Frankfurt gelungen, mittels Kryo-Elektronenmikroskopie (Kryo-EM) die dreidimensionale Struktur von TSEN im Komplex mit einer prä-tRNA aufzuklären.
Anhand ihrer Kryo-EM-Rekonstruktionen, welche mit Hilfe von Infrastruktureinrichtungen der Julius-Maximilians-Universität Würzburg und des Instituts für Biochemie der Goethe-Universität Frankfurt erstellt wurden, konnte das Wissenschaftsteam erstmals zeigen, wie TSEN an die L-förmige prä-tRNA bindet. Aus dem langen Arm des L schneidet TSEN dann das Intron heraus. „Zunächst lagert sich TSEN in den Knick des L ein. Es kann dann sowohl den kurzen und den langen Arm, als auch den Winkel zwischen den Armen erkennen“, erklärt Trowitzsch.
Dabei spielt eine TSEN-Untereinheit (TSEN54) eine herausragende Rolle, wie die Forscher nun beweisen konnten. Die Untereinheit dient als „molekulares Lineal“ und misst den Abstand zwischen dem langen und dem kurzen Arm des L. Auf diese Weise erkennt TSEN, an welcher Stelle die prä-tRNA geschnitten werden muss, um das Intron zu entfernen.
Eine Überraschung waren neue Erkenntnisse zur Interaktion der RNA-Kinase CLP1 und der TSEN-Untereinheit TSEN54: Offensichtlich bindet CLP1 an einen unstrukturierten und dadurch sehr flexiblen Bereich von TSEN54. Exakt dieser Bereich von TSEN54 beinhaltet eine Aminosäure, die bei den meisten Patienten mit Pontozerebellärer Hypoplasie verändert ist. „Für uns ist das ein entscheidender Hinweis darauf, dass sich die Wirkstoffentwicklung zukünftig auf die Aufrechterhaltung der Interaktion von TSEN und CLP1 konzentrieren sollte“, ist Samoil Sekulovski, Erstautor der Studie, überzeugt.
Die Hoffnung ist nun, dass es die Strukturdaten erlauben, Modelle für die Suche nach potenziellen Wirkstoffen zu simulieren. So resümiert Trowitzsch: „Zwar liegt damit immer noch ein sehr weiter Weg bis zu einer erfolgsversprechenden Therapie vor uns, doch unsere Strukturen bilden einen soliden Grundstein, um die Funktionsweise von TSEN und die Krankheitsbilder ihrer Mutanten besser zu verstehen.“
Publikation: Samoil Sekulovski, Lukas Sušac, Lukas S. Stelzl, Robert Tampé, Simon Trowitzsch: Structural basis of substrate recognition by human tRNA splicing endonuclease TSEN. Nature Structural & Molecular Biology (2023) https://doi.org/10.1038/s41594-023-00992-y
News&Views: Anita K. Hopper & Jinwei Zhang: Captured: the elusive eukaryotic tRNA splicing enzyme. Nature Structural & Molecular Biology (2023) https://doi.org/10.1038/s41594-023-00995-9