Volker Zickermann und Eric Helfrich sind bei der Exzellenzcluster-Initiative SCALE (Subcellular Architecture of Life) dabei und werden dort ihre Expertise einbringen. Das Spezialgebiet des einen ist ein Proteinkomplex in den Mitochondrien, den Kraftwerken der Zelle. Der andere sucht schwerpunktmäßig bisher unbekannte Naturstoffe, die die Basis für neue Antibiotika sein könnten.
Auch die Biologie hat Dunkle Materie
Bakterien, Pilze oder Pflanzen produzieren bestimmte Substanzen, um miteinander zu kommunizieren oder Feinde abzuwehren. Diese Naturstoffe sind für die Medizin unverzichtbar: Mehr als 50 Prozent aller zugelassenen Medikamente basieren darauf oder wurden zumindest von ihnen inspiriert. Bakterien sind besonders wichtige Naturstoff-Produzenten, stammen die meisten Antibiotika zur Bekämpfung bakterieller Infektionen doch aus Bakterien. Jedoch gibt es ein Riesenproblem: Antibiotika-Resistenzen verbreiten sich immer weiter und immer schneller. „Wenn wir nicht gegensteuern, sterben in Zukunft viele Menschen an bakteriellen Infektionen, da keines unserer Antibiotika mehr gegen multiresistente Keime wirksam ist“, so Eric Helfrich vom LOEWE-Zentrum für Translationale Biodiversitätsgenomik. Der Leiter der Abteilung Naturstoffgenomik will helfen, dieses „Horror-Szenarium“ abzuwenden, indem er mit seinem Team bisher unbekannte bakterielle Naturstoffe ausfindig macht. Dafür verfolgt er einen Computer-gestützten Ansatz, Genome Mining. Seine Gruppe entwickelt Machine-Learning-Algorithmen, die die Genomsequenzen von Bakterien analysieren und so deren Potenzial zur Naturstoffherstellung bestimmen. Die Naturstoffe könnten die Leitstrukturen für die Entwicklung künftiger Medikamente zur Bekämpfung von Infektionskrankheiten sein.
Grundsätzlich ist das „Schürfen“ im Bakterien-Genom verhältnismäßig leicht, erklärt Helfrich. „Es gibt da bestimmte Gene, die für Enzyme kodieren, welche das Grundgerüst des Naturstoffs aufbauen und modifizieren. Bei Pflanzen sind diese Gene mosaikartig über das ganze Genom verstreut – das erschwert die Identifizierung. In Bakterien liegen sie dagegen direkt nebeneinander vor und bilden Biosynthese-Gencluster. Solche Cluster suchen wir – und zwar diejenigen, die von bisher eingesetzten Algorithmen übersehen wurden.“ Dafür entwickelt das Helfrich-Team zunächst Hypothesen, welche Art Gencluster bisher durchs Suchraster gerutscht sind und trainiert dann die Algorithmen, genau diese zu identifizieren. „So machen wir die biosynthetische Dunkle Materie zugänglich, also die bisher unentdeckten Cluster, die als Bauplan für die Herstellung von Naturstoffen dienen.“
Eine Schwierigkeit bei der Suche nach neuen Naturstoffen liegt darin, dass die meisten Cluster unter Laborbedingungen „stillgelegt“ sind, also keinen Naturstoff produzieren. Viele Vertreter der Streptomyceten etwa können 30 oder 40 unterschiedliche Naturstoffe herstellen, aber selbst bei den am besten untersuchten Organismen sind meist nur eine Handvoll bekannt. Um unentdeckte Cluster zu finden, wird im Helfrich Lab ein Trick angewendet. „Wenn wir mit unseren Algorithmen interessante Cluster gefunden haben, optimieren wir sie und bringen sie dann in geeignete Wirtsorganismen ein. So lässt sich das stille Cluster in den meisten Fällen wieder aktivieren.“
Alle möglichen Bakterien werden untersucht, entscheidend ist aber, welche Art Stoff gefunden werden soll. Aktuell konzentriert sich die Arbeit auf ribosomal synthetisierte und posttranslational modifizierte Peptide. Im Vergleich zu vielen anderen Naturstoffklassen sind diese „RiPPs„ eine heterogene Naturstoffklasse. Es gibt viele verschiedene RiPP-Familien, die bis auf ihre biosynthetischen Grundprinzipien meist nichts miteinander gemein haben. Das macht es mit existierenden Genome Mining Tools fast unmöglich, neue RiPP-Familien zu identifizieren. Ein im Helfrich Lab entwickelter Algorithmus ist jedoch in der Lage, sie aufzuspüren. „Bei der Biosynthese von RiPPs ist es so, dass zunächst ein Vorläufer-Peptid hergestellt wird, welches bestimmte Enzyme modifizieren. Das Peptid lässt sich dann teilweise auf den ersten Blick gar nicht mehr als Peptid identifizieren. Unser Algorithmus erkennt aber den speziellen molekularen Fingerabdruck der Enzyme, die Peptide modifizieren. So finden wir die Peptid-Biosynthese-Gencluster doch – und damit auch neue bakterielle Naturstoffe.“ Das Verfahren ist auch auf andere Naturstoffklassen anwendbar, etwa Terpene. Terpene kennen viele wegen ihrer charakteristischen Gerüche. So ist der Geruch nach Sommerregen auf das Terpen Geosmin aus Bodenbakterien zurückzuführen. Terpene als Naturstoffe sind vielversprechend: Sie besitzen exzellente physisch-chemische Eigenschaften, wie sie sonst nur synthetische Wirkstoffe haben.
Hat der Algorithmus ein aussichtsreiches Cluster gefunden, wird der dazugehörige Naturstoff isoliert und als Nächstes geprüft, ob er tatsächlich in der Lage ist, multiresistente Krankenhauskeime zu töten. „Wir beobachten die Bioaktivität zuerst in einem künstlichen Nährmedium, dann schicken wir den Naturstoff an unsere Kooperationspartner, die ihn an Maus-Infektions-Modellen testen.“ Im Helfrich Lab wurden schon einige Naturstoffe aufgespürt, die gegen multiresistente Keime, aber auch gegen Pilz- oder Krebserkrankungen wirksam sind. „Leider ist es ein weiter Weg bis zur Marktreife“, so Helfrich. „Das kann 15 Jahre dauern und ist extrem teuer, weswegen wir auf die Ressourcen von Pharmaunternehmen angewiesen sind, die auf unsere Forschung aufmerksam werden.“
Die L-förmige Protonenpumpe
In unserem Körper werden mit der Nahrung aufgenommene energiereiche Moleküle, darunter Glucose, schrittweise abgebaut und dienen letztlich der Produktion von Adenosintriphosphat (ATP), dem molekularen Treibstoff der Zelle. Auf dem Weg dahin spielt die Atmungskette eine wesentliche Rolle. Schauplatz dieses Stoffwechselprozesses sind die Mitochondrien, genauer die innere Membran der Zellorganellen. Denn Mitochondrien haben eine äußere Membran und eine innere, welche durch Einstülpungen gefaltet ist. Dazwischen liegt der Zwischenmembranraum. Und ganz innen, von der inneren Membran umschlossen, befindet sich die Matrix.
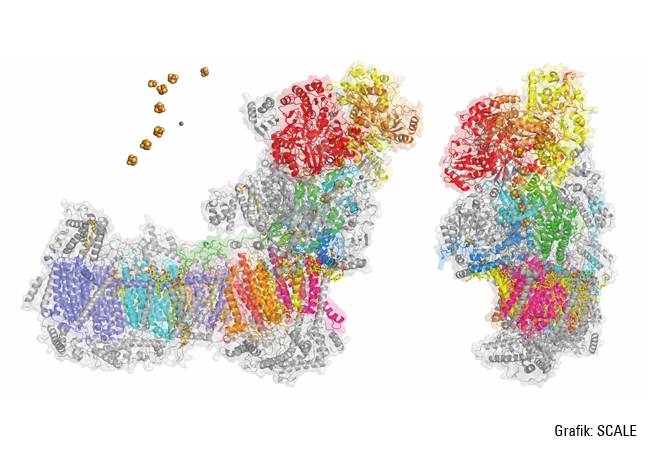
Die Atmungskette besteht aus vier Proteineinheiten, den Komplexen I bis IV. Volker Zickermann vom Institut für Biochemie II der Uniklinik erforscht Komplex I. Hier wird das vorher unter anderem beim Glucose-Abbau gebildete Molekül NADH oxidiert, es gibt Elektronen ab. Und das Molekül Ubichinon („Q10“) wird reduziert, nimmt Elektronen auf. Die bei dieser Redoxreaktion freigesetzte Energie wird dafür genutzt, positiv geladene Wasserstoffteilchen, Protonen, durch die innere Mitochondrienmembran in den Zwischenmembranraum zu pumpen. So ensteht ein Konzentrationsgefälle – innen wenige, außen viele Protonen. Dieser Gradient treibt das Enzym ATP-Synthase an, das dabei teils wie eine Turbine in eine Drehbewegung versetzt wird. „Komplex I ist also eine redoxgetriebene Protonenpumpe“, so Zickermann. „Sie steuert ungefähr 40 Prozent der protonenmotorischen Kraft bei, die zur ATP-Synthese, der Bildung des Energieträgers ATP, nötig ist.“
Die Form von Komplex I ist ganz charakteristisch: ein L mit einem Arm, der in die mitochondriale Matrix ragt, und einem Arm, der in die innere Mitochondrienmembran eingebettet ist. „Alle Redoxreaktionen laufen im Matrixarm ab, das Protonenpumpen wird vom Membranarm bewerkstelligt. Die beiden Aktivitäten sind also räumlich getrennt, aber gekoppelt. Wie genau die Kopplung abläuft, ist immer noch unklar.“ Zickermann will die Mechanismen, die bei Aufbau und Funktionsausübung der molekularen Maschine involviert sind, vollständig aufklären. Dafür untersucht er ihn in Zusammenarbeit mit dem Max-Planck-Institut für Biophysik in Frankfurt mittels Kryo-Elektronenmikroskopie – und profitiert dabei von den großen technischen Fortschritten, die es in den letzten Jahren gab. „Wir können die Struktur von Komplex I mittlerweile in einer Auflösung abbilden, bei der sogar Wassermoleküle im Membranarm zu erkennen sind. Solche Auflösungen ermöglichen Simulationen, die auch die Dynamik in Komplex I sichtbar machen.” Neben den passenden Verfahren braucht es den passenden Modellorganismus: den Hefepilz Yarrowia lipolytica, der unter anderem zur Käsereifung eingesetzt wird. Diese Hefe ist genetisch gut zugänglich und hat einen Komplex I. Unter anderem mit Yarrowia lipolytica als Test-Organismus konnten Zickermann und sein Team Teilschritte der Biogenese von Komplex I bereits klären. „Er hat mehr als 40 Untereinheiten, und diese Bausteine müssen alle an die richtige Stelle gesetzt werden, damit die Protonenpumpe funktioniert. Das erledigen Hilfswerkzeuge wie NDUFAF1, sogenannte Assemblierungsfaktoren.“
Zickermann fand auch heraus, dass ein bestimmtes Protein, Tafazzin, an der Biogenese von Komplex 1 in der Hefe beteiligt ist. „Eine überraschende Entdeckung, denn Tafazzin kennen wir sonst aus einem anderen Zusammenhang. Es ist für das sogenannte Remodelling des Lipids Cardiolipin zuständig.“ Cardiolipin kommt in Eukaryoten praktisch nur in den Mitochondrien vor und ist sehr wichtig für den Aufbau der inneren Mitochondrienmembran. Es ermöglicht vermutlich die starke Krümmung der Cristae, also der Ausstülpungen der inneren Membran. Funktioniert es nicht, leiden Menschen unter dem Barth-Syndrom. Diese Krankheit kann unter anderem zu Herzproblemen und Muskelschwäche führen. „Die Ergebnisse von Studien mit Yarrowia lipolytica lassen sich zwar auf den Menschen übertragen, aber nur in gewissen Grenzen“, ordnet Zickermann ein. Der Assemblierungsfaktor NDUFAF1 kommt auch bei uns vor. Tafazzin ebenso, jedoch nicht in Komplex I.
Der Forscher möchte noch mehr Details über den Gesamtprozess der Biogenese klären. Dabei gibt es die Schwierigkeit, dass Komplex-I-Untereinheiten von zwei unterschiedlichen Genomen kodiert werden, dem Genom im Zellkern und dem mitochondrialen Genom. Es ist unklar, wie die Koordination der beiden Genome funktioniert.
Komplex I ist für den Energiestoffwechsel essenziell, sagt Zickermann. Mutationen können mitochondriale Erkrankungen verursachen. Die Erbkrankheit Lebersche Hereditäre Optikus-Neuropathie (LHON) zum Beispiel, die zu Sehverlust oder Erblindung führen kann. „Nur wenn wir die Mechanismen in Komplex I vollständig verstehen, können wir auch die Pathogenese von LHON und anderen Krankheiten verstehen. Also warum ein Mensch überhaupt daran erkrankt.“
Welche Pläne haben die Forscher bei SCALE?
Neben der Erzeugung von nicht-natürlichen Naturstoffen, die als intelligente Proben bei der Erforschung der subzellulären Architektur von Zellen verwendet werden, will Helfrich seine Expertise auch zur Erforschung von subzellulären Kompartimenten und der Stresstoleranz der bakteriellen Zellhülle einsetzen. „Wer sagt, dass wir nur nach Naturstoffen schürfen können? Die gleichen Prinzipien lassen sich auch auf viele andere spannende Fragestellungen anwenden, wie es sie bei SCALE reichlich gibt.“ Volker Zickermann untersucht auch im Rahmen von SCALE die Vorgänge in Komplex I. „Konkret geht es darum, wie die Biogenese von Komplex I und ähnlichen Komplexen mit der Struktur der Cristae-Membran zusammenhängt, also Einstülpungen der gefalteten inneren Mitochiondrienmembran.“ Zum Beispiel, wo und wann welcher Teilschritt erfolgt und wie der Prozess koordiniert wird.
Andreas Lorenz-Meyer