Neue Methoden zu entwickeln, um bisher unentdeckten Naturstoffen auf die Spur zu kommen – dieses Ziel hat sich Eric Helfrich gesetzt, Professor für Naturstoffgenomik am hessischen LOEWE-Zentrum für Translationale Biodiversitätsgenomik (LOEWE-TBG) und der Frankfurter Goethe-Universität. Für seine Forschung erhält er seit Januar 2023 eine sechsjährige Förderung im Emmy Noether-Programm der Deutschen Forschungsgemeinschaft (DFG) für herausragend qualifizierte Nachwuchswissenschaftler*innen in Höhe von rund 1,5 Millionen Euro. Das Besondere an seinem Ansatz: Die genetischen Baupläne, die für die Produktion unbekannter Naturstoffe verantwortlich sind, werden mittels Maschinellem Lernen im Erbgut von Organismen ausfindig gemacht und anschließend mit neuen Verfahren der Synthetischen Biologie in Bakterien biotechnologisch hergestellt.

Zahlreiche Lebewesen, darunter Pflanzen, Tiere, Pilze und Bakterien, produzieren für die Wechselwirkungen mit ihrer Umwelt Naturstoffe – von denen auch wir Menschen profitieren, sei es als Medikamente, in der Landwirtschaft oder in der Lebensmittelindustrie. Einen Meilenstein für ihre pharmazeutische Nutzung bildete vor knapp einhundert Jahren, 1928, die Entdeckung des Antibiotikums Penicillin durch Alexander Fleming. Inzwischen beruht etwa die Hälfte aller zugelassenen Wirkstoffe für Medikamente auf Naturstoffen. Um neue Substanzen zu finden und sie passgenau nutzen zu können, analysieren Forschende weltweit, wie Naturstoffe gebildet werden und durch welche Eigenschaften und Wirkung sie sich auszeichnen. Für die unterschiedlichen Naturstoffklassen wurden dabei jeweils charakteristische Grundsätze ihrer Herstellung identifiziert. Inzwischen sind jedoch auch Naturstoffe bekannt, die diesen scheinbar universellen Prinzipien nicht folgen. Entsprechend werden sie bei der computergestützten Suche nach ungewöhnlichen genetischen Bauplänen für die Produktion der Substanzen mit den derzeit verfügbaren bioinformatischen Algorithmen nicht detektiert.
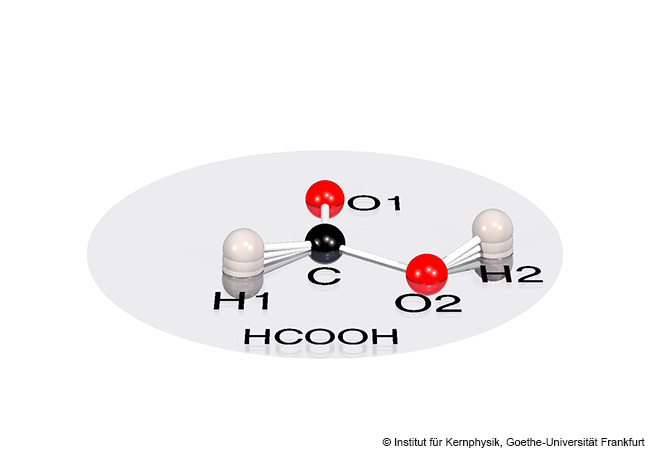
Für den Frankfurter Naturstoffgenomiker Prof. Dr. Eric Helfrich ist diese Erkenntnis Anlass, auf Spurensuche zu gehen. Sein Schwerpunkt liegt im sogenannten Genome-Mining für Naturstoffe – dem Auffinden neuer Substanzen durch genaue Analyse der für ihre Produktion zuständigen Bereiche im Erbgut von Organismen. Dabei konzentriert er sich auf Naturstoffe aus Bakterien, die potenziell in der Medizin unter anderem gegen Infektionserkrankungen verwendet werden könnten. Mit seinem neuen von der DFG geförderten Emmy Noether-Projekt „Peptid Biosynthese abseits bekannter Wege: Machine Learning-basierte Identifizierung ungewöhnlicher Peptid Naturstoffe“ richtet er den Blick auf Peptide als kleine Eiweiße, die durch die Verknüpfung von einzelnen Aminosäuren entstehen. Einige dieser Peptide werden in der Human- und Tiermedizin bereits als Antibiotika oder auch als Medikamente gegen Krebserkrankungen eingesetzt. In der Lebensmittelindustrie dienen sie beispielsweise als Konservierungsstoff.
„Es sind bereits zahlreiche Peptid-Naturstoffe bekannt und gut erforscht, und sie spielen eine wichtige Rolle in der Medizin und bei der Produktion von Lebensmitteln. Doch wir sind uns sicher, dass es noch sehr viel mehr nützliche Peptide zu entdecken gibt, wenn wir die Grenzen bisheriger Denkmuster erweitern“, betont Helfrich. Dafür will er mit den Projektmitteln neue bioinformatische Algorithmen entwickeln, um gezielt Baupläne für die Herstellung von Peptiden – sogenannte biosynthetische Gencluster – in Bakterien zu identifizieren, die nicht den bisher bekannten Schemata entsprechen. „Dabei setzen wir in den Analyseprozessen auf Maschinelles Lernen“, so Helfrich. Grundlage dafür bilden zunächst vom Team aufgestellte Hypothesen. Diese werden dann durch Maschinelles Lernen bestätigt, indem bestimmte Muster identifiziert werden, die das menschliche Auge allein nicht erkennen könnte. Die entwickelten Algorithmen werden dazu verwendet, ungewöhnliche genetische Baupläne für die Herstellung von Peptiden in hunderttausenden von Bakteriengenomen zu finden. Vielversprechende Baupläne werden daraufhin mittels moderner Methoden der Synthetischen Biologie in Bakterien eingebracht, um die gewünschten Peptide biotechnologisch herzustellen. Dank der Emmy Noether-Förderung kann Helfrich ein neues Massenspektrometer anschaffen, um die produzierten Naturstoffe nachweisen und charakterisieren zu können, und sein Team erweitern.
„Unsere Forschung wird dazu beitragen, Licht in bislang unbekannte Wege der bakteriellen Peptid-Herstellung zu bringen und damit das bisherige Lehrbuchwissen zu erweitern“, beschreibt Helfrich das Ziel seiner Forschung. „Dabei wollen wir natürlich auch neue Substanzen entdecken, die künftig medizinisch genutzt werden können. Dafür arbeiten wir eng mit Partner*innen innerhalb von LOEWE-TBG zusammen, die die Naturstoffe auf ihr pharmazeutisches Potential hin untersuchen.“
Helfrichs Forschung zum Genome Mining ist am LOEWE-Zentrum TBG im Bereich „Naturstoffgenomik“ angesiedelt, in dem die beteiligten Forscher*innen unterschiedliche Facetten von Naturstoffen und Artenvielfalt analysieren, von Tiergiften über Flechten bis zu Bakterien. Dabei haben sie immer auch das Anwendungspotenzial der Ergebnisse im Blick. An der Goethe-Universität Frankfurt forscht Helfrich am Institut für Molekulare Biowissenschaften und vermittelt in der Lehre unter anderem die große Bedeutung von Naturstoffen, die sie mit ihren wichtigen ökologischen und pharmazeutisch nutzbaren Eigenschaften einnehmen. Die Studierenden erhalten dabei Einblick in neue Methoden aus den Bereichen Informatik, Molekularbiologie, Synthetische Biologie und Analytische Chemie. Diese Interdisziplinarität in der Forschung ermöglicht es erst, neuartige Naturstoffe ausfindig zu machen.
Quelle: Pressemitteilung LOEWE-TBG