Kühe können sich an Nahrung mit unterschiedlichem Kochsalzgehalt anpassen. Wie sie das machen, war bislang ein Geheimnis. Jetzt haben Forscher der Goethe-Universität im Mikrobiom des Pansens ein Bakterium entdeckt, das einen neuen Typ von Zellatmung hat.
Die Kuh kann Gras nur mithilfe von Milliarden Mikroorganismen in ihrem Pansen verwerten. Ein ganzer Zoo von Bakterien, Archaeen und Protozoen arbeitet dort wie am Fließband: Zuerst spalten diese Einzeller die Cellulose, einen Vielfachzucker, auf. Andere Bakterien vergären die freigesetzten Zucker zu Fettsäuren, Alkoholen und Gasen wie Wasserstoff und Kohlendioxid. Schließlich verwandeln methanogene Archaeen diese beiden Gase zu Methan.
Eine durchschnittliche Kuh produziert etwa 110 Liter Methan pro Tag. Durch das Wiederkäuen entweicht es aus dem Maul, wird aber auch wieder mit Nahrungsbrei und vermischt. Dadurch kann der Kochsalzgehalt des Grasbreis stark schwanken (zwischen 60 – 800 milli-Mol Natriumchlorid (NaCl) pro Liter).
Wie sich die Bakterien des Pansens an diese stark schwankenden Kochsalzkonzentrationen anpassen, hat eine deutsch-amerikanische Forschergruppe jetzt herausgefunden:„Bioinformatische Analysen der Genome von Pansenbakterien führten unseren amerikanischen Kollegen Tim Hackmann zu der Vermutung, dass einige Pansenbakterien zwei unterschiedliche Atmungsketten haben. Eine davon funktioniert mit Natriumionen, die andere ohne“, erklärt Prof. Volker Müller von der Abteilung Molekulare Mikrobiologie und Bioenergetik an der Goethe-Universität. Müller schlug seiner Doktorandin Marie Schölmerich deshalb vor, einen typischen Vertreter im Mikrobiom von Wiederkäuen zu untersuchen: das Bakterium Pseudobutyrivibrio ruminis.
Marie Schölmerich hat zusammen mit der Bachelorstudentin Judith Dönig und dem Masterstudenten Alexander Katsyv das Bakterium kultiviert. Tatsächlich konnten sie beide Atmungsketten nachweisen. Wie die Forscher in der aktuellen Ausgabe der Proceedings of the National Academy of Sciences (PNAS) berichten, wird während der Zuckeroxidation der Elektronenüberträger Ferredoxin (Fd) reduziert. Reduziertes Ferredoxin treibt beide Atmungsketten an.
Die eine Atmungskette besteht aus dem Enzymkomplex Fd:NAD-Oxidoreduktase (Rnf- Komplex). Er transportiert Natriumionen unter Aufwendung von Energie aus der Zelle. Bei ihrem Wiedereintritt treiben die Natriumionen eine ATP-Synthase an, so dass ATP entsteht. Diese Atmungskette arbeitet nur in Gegenwart von Natrium-Ionen.
Fehlen Natrium-Ionen, bildet das Bakterium eine alternative Atmungskette mit einem anderen Enzymkomplex: Die Ech-Hydrogenase oder synonym Fd:H+-Oxidoreduktase produziert Wasserstoff und pumpt Protonen aus der Zelle. Treten diese über eine zweite ATP-Synthase, die Protonen, aber keine Natriumionen akzeptiert, wieder in die Zelle ein, entsteht ebenfalls ATP.
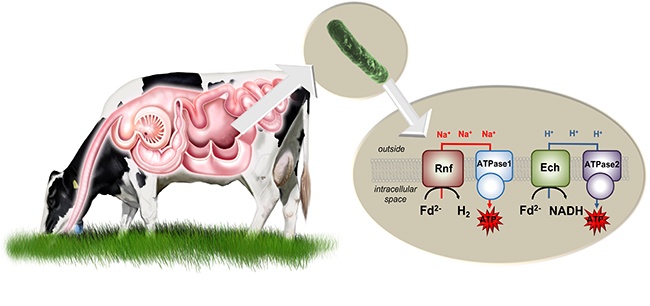
Das Bakterium Pseudobutyrivibrio ruminis (grün), ein typisches Pansenbakterium, gewinnt Energie über zwei unterschiedliche Atmungsketten. Die eine benötigt Natriumionen, die andere Wasserstoffionen (H+). So passt es sich optimal an die schwankende Kochsalzkonzentration der Nahrung an. (Grafik: Goethe-Universität/ Kuh: Shutterstock)
„Bis heute ist dies das erste Bakterium, bei dem diese beiden einfachen, komplett unterschiedlichen Atmungsketten nachgewiesen wurden. Unsere bioinformatischen Analysen legen aber nahe, dass sie auch bei anderen Bakterien zu finden sind“, erklärt Marie Schölmerich. „Diese Anpassungsstrategie scheint also weiter verbreitet zu sein“, lautet ihre Vermutung.
Interessanterweise wurden die beiden Enzymkomplexe (Rnf-und Ech-Komplex) auch in evolutionsbiologisch alten Bakterien gefunden. Die Arbeitsgruppe von Prof. Müller hat sie eingehend untersucht, aber immer nur einen der beiden Enzymkomplexe gefunden, nie beide zusammen. „Jetzt werden wir mit Methoden der synthetischen Mikrobiologie Hybride von Bakterien herstellen, die beide Komplexe enthalten, um diese für biotechnologische Prozesse zu optimieren. Dadurch kann man den zellulären ATP-Gehalt erhöhen. Dann lassen sich wertvollere Produkte herstellen“, erklärt Prof. Müller. Geplant ist, die Atmungsketten einzusetzen, um durch die Fermentation von Synthesegasen Wertstoffe zu gewinnen. Dies ist Gegenstand der Untersuchungen in einem BMBF-geförderten Projekt.
Publikation: Schölmerich, M.C., Katsyv, A., Dönig, J., Hackmann, T., Müller, V. (20XX). Energy conservation involving two respiratory circuits. Proc. Natl. Acad. Sci. U.S.A., https://doi.org/10.1073/pnas.1914939117.