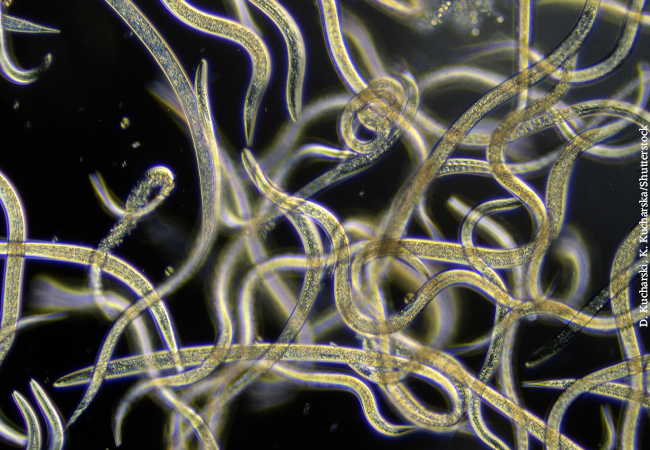
In der Natur leben häufig verschiedene Organismen miteinander in Symbiose – und profitieren damit voneinander. Dies trifft auch auf bestimmte Fadenwürmer zu, die gemeinsam mit Bakterien ihre Beute „erlegen“. Sobald die Fadenwürmer in ihr Opfer, vorzugsweise Insektenlarven, eingedrungen sind, übernehmen die Bakterien aus ihrem Darm die Regie und töten die Larve mit giftigen Substanzen. Hilfreicher Nebeneffekt: Die so vergiftete Larve wird von Konkurrenten bei der Nahrungssuche verschmäht. Ein Team von Wissenschaftler*innen aus Frankfurt am Main und Jena identifizierte nun einen Regulator, der die bei diesem Beutezug ablaufenden biologischen Prozesse maßgeblich beeinflusst. Die gemeinsame Publikation ist online in der Fachzeitschrift „Nature Microbiology“ veröffentlicht.
Die Bakterien, die die Fadenwürmer mit ihrem Gift unterstützen, entstammen den Gattungen Photorhabdus und Xenorhabdus. Gemeinsam bilden sie eine sogenannte obligatorische Symbiose; beide Partner sind aneinander gebunden und können in der Natur nicht ohne den anderen existieren. Während der Fadenwurm von der Giftbildung seines Bakteriums profitiert, erhält das Darmbakterium einen geschützten und passenden Lebensraum. Dort produziert es dann eine komplexe Mischung verschiedener Moleküle, sogenannte Sekundärmetabolite, die in diesem Fall giftig wirkt.
Sekundärmetabolite spielen auch in der Humanmedizin eine wichtige Rolle. So basieren zum Beispiel viele der klinisch genutzten Antibiotika und Krebsmedikamente auf bakteriellen Sekundärmetaboliten.
Wie die Bildung dieses Sekundärstoff-Cocktails reguliert wird, ist zum Teil bereits erforscht – nicht jedoch, welche Rolle kleinen Ribonukleinsäuren (sRNA) dabei zukommt. Diese existieren in nahezu allen Mikroorganismen und übernehmen hier eine wichtige Funktion bei der Kontrolle der Proteinproduktion. Die an der Studie beteiligten Wissenschaftler*innen konnten nun eine entscheidende Beteiligung dieser sRNAs an der Produktion der Sekundärmetabolite nachweisen: Die als „ArcZ“ bezeichnete sRNA beeinflusst etwa 15 Prozent aller Gene der Bakterien – darunter alle Gene, die an der Bildung der Sekundärmetabolite mitwirken.
„Die Produktion der Sekundärmetabolite scheint auch in vielen anderen Bakteriengattungen abhängig von ArcZ zu sein“, erklärt Helge Bode, Professor für Molekulare Biotechnologie an der Goethe-Universität Frankfurt und am LOEWE-Zentrum für Translationale Biodiversitätsgenomik (TBG). „Durch die Aufklärung des zugrundeliegenden Mechanismus können wir nun auch in diesen Bakterien gezielt neue Sekundärmetabolite produzieren, die möglicherweise in Zukunft auch klinisch eingesetzt werden können“, ergänzt Nick Neubacher, Doktorand an der Goethe-Universität Frankfurt und Erstautor der Publikation.
„Unsere Arbeit verknüpft damit zwei Forschungsgebiete, die bisher nahezu keine Berührungspunkte haben: die Forschung an regulatorischen RNAs und an Sekundärmetaboliten“, erläutert Kai Papenfort, Professor für Allgemeine Mikrobiologie an der Friedrich-Schiller-Universität Jena und am dortigen Exzellenzcluster „Balance of the Microverse“.
So ist diese Forschung eine enge Teamarbeit von Wissenschaftler*innen verschiedener Disziplinen und wurde neben der Federführung im Rahmen des Sonderforschungsbereichs 902 „Molecular Principles of RNA-based Regulation“ der Deutschen Forschungsgemeinschaft unter anderem auch vom Exzellenzcluster 2051 „Balance of the Microverse“ und dem LOEWE-Zentrum für Translationale Biodiversitätsgenomik (TBG) unterstützt.
Publikation: Nick Neubacher, Nicholas J. Tobias, Michaela Huber, Xiaofeng Cai, Timo Glatter, Sacha J. Pidot, Timothy P. Stinear, Anna Lena Lütticke, Kai Papenfort and Helge B. Bode: Symbiosis, virulence and natural-product biosynthesis in entomopathogenic bacteria are regulated by a small RNA. https://www.nature.com/articles/s41564-020-00797-5
Diese Meldung ist zuerst bei Senckenberg Gesellschaft für Naturforschung erschienen.