Noch detailliertere Einblicke in die Zelle sind künftig möglich mit Hilfe einer Neuentwicklung, an der die Goethe-Universität beteiligt war: Der Arbeitsgruppe um Prof. Harald Schwalbe ist es gemeinsam mit Wissenschaftlern aus Israel gelungen, die Kernspinresonanz (NMR)-Methode zur Untersuchung von RNA hunderttausendfach zu beschleunigen.
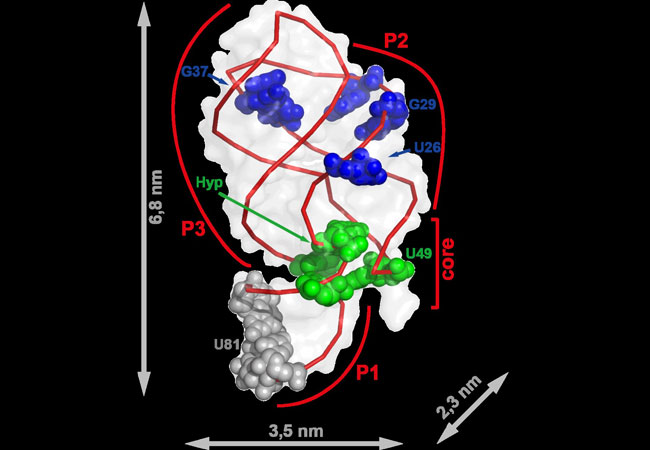
Wie ein einzelnes Puzzlestück sich in ein Puzzle einfügt, so bindet das Molekül Hypoxanthin an eine Ribonukleinsäure(RNA)-Kette, die daraufhin innerhalb einer Sekunde ihre dreidimensionale Gestalt verändert und so neue Prozesse in der Zelle in Gang setzt. Kaum vorstellbar winzige Strukturveränderungen in Zellen können Wissenschaftler nun dank einer verbesserten Methode verfolgen – und zwar sowohl in ihrem zeitlichen als auch räumlichen Ablauf. Der Arbeitsgruppe um Harald Schwalbe vom Zentrum für Biomolekulare Magnetische Resonanz der Goethe-Universität ist es zusammen mit Wissenschaftlern aus Israel gelungen, die Kernspinresonanz(NMR)-Methode zur Untersuchung von RNA auf das Hunderttausendfache zu beschleunigen.
„Dies erlaubt uns erstmalig, die Dynamik von RNA-Strukturänderungen so schnell zu verfolgen, wie sie auch in der Zelle ablaufen“, beschreibt Schwalbe diesen wissenschaftlichen Durchbruch und betont: „Das Team um Lucio Frydmann vom Weizmann-Institut in Israel hat dazu wesentlich beigetragen.“
Die neuartige NMR-Untersuchungen nutzen Wassermoleküle, deren Atome sich in einem Magnetfeld verfolgen lassen. Schwalbe und sein Team erzeugen hyperpolarisiertes Wasser. Dazu setzen sie dem Wasser eine Verbindung zu, die permanent ungepaarte Elektronen-Radikale besitzt. Durch Anregung mit einer Mikrowelle bei -271°C kann man die Elektronen im Magnetfeld ausrichten. Diese unnatürliche Ausrichtung erzeugt eine Polarisation, die auf Polarisation der im NMR eingesetzten Wasserstoffatome bei +36°C übertragen wird. Das so polarisierte Wassermolekül erwärmen sie in wenigen Millisekunden und geben es zusammen mit Hypoxanthin auf die RNA-Kette. Damit können sie auch schnelle chemische Reaktionen und Faltungsänderungen von Biomolekülen auf Atom-Ebene beobachten.
Die Imino-Gruppen in RNA lassen sich mit dieser Methode besonders gut analysieren. Die Wissenschaftler konnten so RNA-Strukturänderungen präzise messen. Sie verfolgten ein kurzes RNA-Stück (Aptamer) aus Bacillus subtilis, das seine Struktur bei der Bindung von Hypoxanthin so ändert, dass die folgenden Boten-RNA (mRNA) nicht mehr abgelesen wird. Derart kleine Änderungen auf Molekülebene steuern viele Prozesse nicht nur in Bakterien, sondern auch in mehrzelligen Lebewesen bis hin zum Menschen.
Die verbesserte Methodik erlaubt es künftig, RNA-Faltungen in Echtzeit zu verfolgen, sogar, wenn sie weniger als eine Sekunde benötigen. Dies ist unter physiologischen Bedingungen möglich, also in flüssiger Umgebung und in natürlicher Molekül-Konzentration bei Temperaturen um 36 Grad Celsius. „Der nächste Schritt wird es jetzt sein, nicht nur einzelne, sondern hunderte RNAs zu untersuchen, um die biologisch wichtigen Unterschiede in ihren Faltungsraten bestimmen zu können“, so Boris Fürtig aus der Arbeitsgruppe Schwalbe.
Publikation: Mihajlo Novakovic, Gregory L. Olsen, György Pintér, Daniel Hymon, Boris Fürtig, Harald Schwalbe, Lucio Frydman: A 300-fold enhancement of imino nucleic acid resonances by hyperpolarized water provides a new window for probing RNA refolding by 1D and 2D NMR, PNAS, 16. Januar 2020 https://doi.org/10.1073/pnas.1916956117