Im Krankenhaus erworbene Infektionen sind oft besonders schwer zu behandeln, weil die Erreger Resistenzen gegen gängige Antibiotika aufweisen. In dieser Hinsicht besonders gefürchtet ist das Bakterium Acinetobacter baumannii, für dessen Bekämpfung unter großem Druck neue Therapieansätze gesucht werden. Bioinformatiker der Goethe-Universität und der Forschergruppe FOR2251 der Deutschen Forschungsgemeinschaft (DFG) haben nun innerhalb von A. baumannii eine unerwartet große Diversität bestimmter Zellanhänge nachgewiesen, die mit der Pathogenität im Zusammenhang stehen. Daraus könnten Behandlungsstrategien resultieren, die spezifisch auf einen bestimmten Keim zugeschnitten sind.
Jährlich erkranken in Europa mehr als 670 000 Menschen an antibiotikaresistenten Erregern, und 33 000 sterben an den von ihnen verursachten Krankheiten. Besonders gefürchtet sind Keime, die unempfindlich gleich gegen mehrere Antibiotika sind. Zu ihnen gehört das Bakterium Acinetobacter baumannii, das heute vor allem als „Krankenhauskeim“ gefürchtet ist: Schätzungen zufolge gehen bis zu fünf Prozent aller im Krankenhaus erworbenen und ein Zehntel aller bakteriellen Infektionen mit tödlichem Ausgang allein auf diesen Keim zurück. Damit steht A. baumannii ganz oben auf einer Liste von Erregern, für die laut der Weltgesundheitsorganisation (WHO) neue Therapien entwickelt werden müssen.
Eine Voraussetzung dafür ist das Verständnis, welche Eigenschaften A. baumannii zu einem Krankheitserreger machen. Bioinformatiker um Prof. Ingo Ebersberger von der Goethe-Universität Frankfurt und dem LOEWE-Zentrum für Translationale Biodiversitätsgenomik (LOEWE-TBG) vergleichen hierzu die Genome und die darin kodierten Proteine über eine Vielzahl unterschiedlicher Acinetobacter-Stämme hinweg. Vor allem aus den Unterschieden zwischen gefährlichen und harmlosen Stämmen können Rückschlüsse gezogen werden, welche Gene zur Pathogenität beitragen.
Entsprechende Studien haben sich mangels geeigneter Methoden bisher darauf konzentriert, ob ein Gen in einem Bakterienstamm vorhanden ist oder nicht. Daneben können Bakterien aber auch neue Eigenschaften erwerben, indem sie schon vorhandene Gene und damit die davon kodierten Proteine verändern. Ebersbergers Team hat deshalb eine bioinformatische Methode entwickelt, um die Veränderung von Proteinen entlang einer evolutionären Linie zu verfolgen und diese Methode nun in Kooperation mit Mikrobiolog:innen des Instituts für Molekulare Biowissenschaften sowie des Instituts für Medizinische Mikrobiologie und Infektionskontrolle der Goethe-Universität erstmals auf Acinetobacter angewendet.
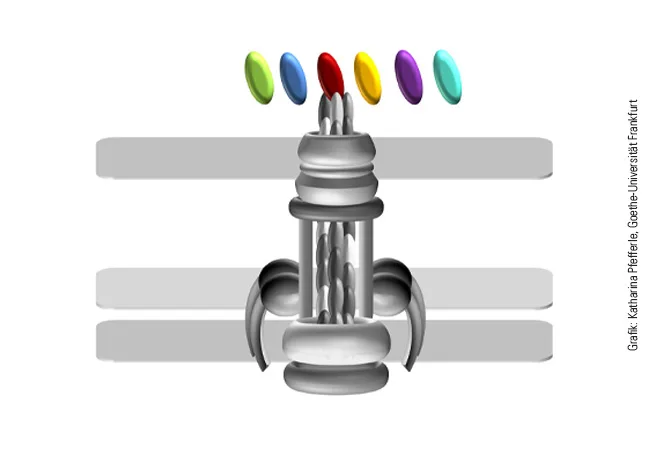
Dabei konzentrierten sich die Forscher:innen auf haarähnliche Zellanhänge, sogenannte Typ-IVa-(T4A)-Pili, die bei Bakterien weit verbreitet sind und die der Interaktion mit der Umwelt dienen. Dass sie einerseits bei ungefährlichen Bakterien vorkommen, andererseits bei manchen Pathogenen sogar als Schlüsselfaktor für die Virulenz identifiziert wurden, deutet darauf hin, dass die T4A-Pili im Laufe der Evolution wiederholt neue, mit der Pathogenität assoziierte Eigenschaften erworben haben.
Tatsächlich konnte das Forschungsteam zeigen, dass das Protein ComC, das an der Spitze der T4A-Pili sitzt und für deren Funktion essenziell ist, innerhalb der Gruppe der pathogenen Acinetobacter-Stämme auffällige Veränderungen zeigt. Selbst verschiedene Stämme von A. baumannii verfügen über unterschiedliche Varianten dieses Proteins. Ebersberger vergleicht daher die T4A-Pili mit einem multifunktionalen Gartenwerkzeug, bei dem der Griff stets gleich bleibt, die Aufsätze aber auswechselbar sind. „Auf diese Weise können über kurze evolutionäre Zeiträume drastische Funktionsänderungen erreicht werden“, ist der Bioinformatiker überzeugt. „Wir gehen davon aus, dass Bakterienstämme, die sich in den T4A-Pili unterscheiden, auch unterschiedlich mit ihrer Umwelt in Kontakt treten. Das kann sich beispielsweise darauf auswirken, in welcher Nische innerhalb des Menschen sich der Erreger ansiedelt.“
Das Wissen um die unerwartet hohe Diversität innerhalb des Keims soll dafür genutzt werden, die Behandlung von Infektionen mit A. baumannii zu verbessern, wie Ebersberger erklärt: „Aufbauend auf unseren Ergebnissen könnten personalisierte Therapien entwickelt werden, die ganz auf einen bestimmten Erregerstamm zugeschnitten sind“. Die Studie von Ebersberger und Kolleg:innen zeigt aber noch etwas anderes: Vermutlich haben bisherige Studien zur vergleichenden Genomik von A. baumannii erst die Spitze des Eisbergs ans Licht gebracht. „Unser Ansatz hat die Auflösung der Suche nach möglichen Komponenten, die Pathogene charakterisieren, deutlich erhöht“, so Ebersberger.
Publikation: Ruben Iruegas, Katharina Pfefferle, Stephan Götting, Beate Averhoff, Ingo Ebersberger: Feature-architecture aware phylogenetic profiling indicates a functional diversification of type IVa pili in the nosocomial pathogen Acinetobacter baumannii. PLOS Genetics (2023) https://doi.org/10.1371/journal.pgen.1010646