Prof. Harald Schwalbe leitete ein internationales Konsortium zur Forschung an SARS-CoV-2. Jetzt wurde es trotz seiner guten Ergebnisse aufgelöst – damit aus Substanzkandidaten Wirkstoffe werden können.
UniReport: Herr Prof. Schwalbe, als Deutschland im März 2020 wegen des Corona-Lockdowns stillstand, starteten Sie ein großes Forschungsprojekt zu SARS-CoV-2 und riefen das COVID-19-NMRKonsortium ins Leben. Die zwischenzeitlich rund 240 beteiligten Forscherinnen und Forscher aus 18 Ländern wollten mit Hilfe der Kernspin-Resonanz-Spektroskopie (NMR) Strukturen des Virus aufklären und damit bestimmen, wie Atome und Moleküle in den Proteinen und im RNA-Erbgut des Virus angeordnet sind. Warum ist es wichtig, das zu wissen?

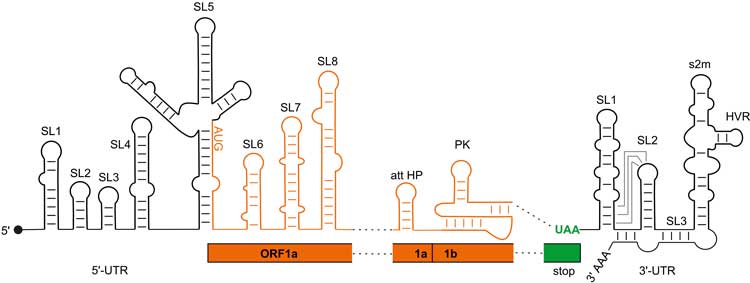
Harald Schwalbe: Wenn man die Struktur des Erregers in allen Details kennt, kann man versuchen, sehr gezielt Moleküle zu finden, die an entscheidenden Stellen der SARS-CoV-2-Proteine oder der Virusgenom-RNA binden und dadurch die Vermehrung des Virus stoppen. Das Virus ist vergleichsweise klein: Es hat nur 27 Proteine und 15 relevante RNA-Abschnitte, die das Infektionsgeschehen steuern. Unser Konsortium hatte sich vorgenommen, zunächst die RNAs sowie die Proteine aufzureinigen und ihre Strukturen über NMR zu bestimmen und dann Substanzbanken kleiner Moleküle danach zu durchsuchen, welche von ihnen an welche Substrukturen der viralen RNAs oder Proteine binden.
Mit Beginn der Pandemie haben sich weltweit Forscherinnen und Forscher intensiv mit SARS-CoV-2 beschäftigt. Um schnelle Erfolge in der Entwicklung von Medikamenten zu haben, fokussierten sich die meisten allerdings auf wichtige Proteine wie das Spike-Protein oder ein Enzym namens Hauptprotease, das zentral für die Reifung der Viruspartikel in der Zelle ist und gegen das auch der Wirkstoff Paxlovir erfolgreich entwickelt wurde. Warum wollten Sie im Konsortium alle RNAs und alle Proteine auf einmal untersuchen?
Seit Beginn des Jahres 2022 sind bisher weltweit Strukturuntersuchungen von 2781 SARS-CoV-2-Proteinen und mehr als 60 RNA-Protein-Komplexen gemacht worden, meist mittels Kristallographie oder hochauflösender Kryo-Elektronentomographie. Doch ein ungeheuer großer Teil dieser Daten ist redundant: 900 der Untersuchungen wurden am Spike-Protein und 500 an der Hauptprotease gemacht. Eine solche Redundanz wollten wir mit Bildung des NMR-Konsortiums vermeiden. Außerdem konnten wir die Präparation der RNAs und der Viren in Laboratorien weltweit standardisieren. Damit sind die Qualität der Daten und die Vergleichbarkeit der Datensätze viel höher, als wenn das von Labor zu Labor immer ein bisschen anders gemacht wird. Gegenüber der Kryo-Elektronentomographie und der Kristallographie hat die NMR zudem den Vorteil, dass man mit ihr auch solche Proteine charakterisieren kann, die dynamisch verschiedene Strukturen annehmen. Insbesondere RNA-Strukturen, mit denen das Virus seine Vermehrung steuert, lassen sich nur über NMR abbilden. Und genau auf RNA-Forschung sind wir hier in Frankfurt spezialisiert: Seit 2000 arbeiten wir in Sonderforschungsbereichen an der Untersuchung von RNAs. Was früher als Nischenforschung angesehen wurde, ist nun in aller Munde: mRNA.
Und heute haben Sie im COVID-19-NMR-Konsortium ihr Ziel erreicht?
Ja, heute habe wir 22 Proteine der 27 SARS-CoV-2-Proteine – fünf hatten wir von vorneherein aus technischen Gründen ausgeschlossen – und alle RNA-Steuerungselemente zugänglich gemacht. Außerdem haben wir eine ganze Reihe an kleinen Molekülen als potenzielle Bindungspartner identifiziert. Viele Kolleginnen und Kollegen haben gesagt: „Aber wir wissen doch, dass das Virus über das Spike-Protein in die Zelle eindringt, und dass die Blockade der Hauptprotease die Virusvermehrung stoppt. Warum fokussiert Ihr Euch nicht auf diese und die wenigen anderen Proteine, die für die Virusinfektion relevant sind?“ Wir haben dagegengehalten, dass wir bislang nicht verstehen, warum zum Beispiel SARSCoV- 2 jetzt weltweit ein viel größeres Problem ist, als es SARS-CoV vor einigen Jahren war. Und dass als Nächstes vielleicht ein Virus namens SARS-CoV-3 kommt. Wir waren daher überzeugt, dass es sich lohnt, SARS-CoV-2 so genau wie möglich anzuschauen. Denn welche Proteine und RNASteuerelemente bei künftigen Mutationen oder neuen, ähnlichen Virusarten relevant sein werden, wissen wir heute noch nicht.
Als Sie 2020 loslegten, herrschten strenge Kontaktbeschränkungen. Wie kann man unter solchen Umständen forschen?
Wir haben in drei Schichten rund um die Uhr gearbeitet: Montagmorgen um 7.30 Uhr haben die ersten beiden Forschenden im Labor angefangen, gearbeitet bis 14.00 Uhr und dann das Labor desinfiziert. Um 14.30 Uhr kam die zweite Schicht rein bis 22.00 Uhr, dann wurde wieder desinfiziert und anschließend startete die Nachtschicht. Mit 30 Leuten konnten wir so alle zwei Wochen vier neue RNA-Proben sauber herstellen, die dann im NMR-Spektrometer vermessen wurden, von anderen in einem zweiten Schichtverfahren. Die NMR-Daten wurden dann in weltweiten Teams digital analysiert. Das war ein Arbeiten wie am Fließband und hat wegen unseres standardisierten Vorgehens hohe Anforderung an die Disziplin gestellt. Funktionieren konnte das nur, weil alle im Team hier in Frankfurt und bei unseren Partnern in Darmstadt extrem motiviert waren. Doch es war auch die Zeit, wo die Alternative darin bestand, zu Hause zu sitzen und sämtliche Netflix-Serien durchzuschauen. Alle, die ich gefragt habe, waren total dankbar, dass sie arbeiten konnten und haben auch die ethische Verantwortung gespürt.
Das Konsortium war sehr erfolgreich – und hat sich jetzt trotzdem aufgelöst. Warum?
Jetzt beginnt eine neue Phase: Wir arbeiten nicht mehr an allen Proteinen und RNAs gleichzeitig, wir fokussieren jetzt alle auf einzelne Proteine oder RNA-Abschnitte. Dafür braucht es keine große Gruppe mehr. Die Arbeiten des Konsortiums haben wir komplett als Open Source auf der Webpage www.covid19-nmr.de allen Forschungsgruppen der Welt zugänglich gemacht und darauf bauen auch wir selber auf. In Frankfurt haben wir mit unseren Kooperationspartnern in Marburg und München und Industriepartnern jetzt zwei Moleküle synthetisiert, die in der Zellkultur die Vermehrung von SARS-CoV-2 sehr erfolgreich hemmen. Damit daraus vielleicht einmal antivirale Medikamente werden können, die ein Unternehmen produzieren und verkaufen kann, haben wir diese Verbindungen patentiert. Wenn die Substanzen einmal in die klinische Forschung gehen sollten, müssen die klinischen Zulassungsphasen am Patienten finanziert werden. So viel Geld investiert nur jemand, der mit einem fertigen Medikament auch Gewinn erwirtschaften kann.
Braucht es also die kleine Gruppe, die »wissenschaftlichen Einzelkämpfer«, genauso wie das in Open Source veröffentlichende Konsortium?
Ja, und das betrifft nicht nur Patente und die Entwicklung von Wirkstoffen, sondern auch die wissenschaftliche Karrie- re. Vor allem in der Grundlagenforschung sind Publikationen mit möglichst wenig Autoren wichtig, damit jemand zeigen kann: Das habe ich erreicht, das qualifiziert mich zum Beispiel für eine Professur. Auf der anderen Seite gibt es aber auch die großen gesellschaftlichen Herausforderungen, wo wir als Wissenschaftlerinnen und Wissenschaftler sozusagen kampagnenfähig sein und eine ganze andere Art von Forschung machen müssen, arbeitsteilig und standardisiert. Hier müssen wir in Deutschland noch schneller werden, um Ressourcen kurzfristig bündeln zu können. Dabei sehe ich die Art, wie große gesellschaftliche Herausforderungen identifiziert werden, durchaus kritisch. Die entsprechenden Forschungsfelder erhalten üppige Finanzierungen, wohinter auch viel Lobbying steckt. Das führt zu Fehlentscheidungen der politischen Entscheidungsträger, die Virologie Jahrzehnte lang als wenig relevant einstuften.

Und wie wird es weitergehen?
Erstmal sind wir dankbar, dass wir im NMR-Konsortium eine bisher nicht bekannte internationale Solidarität im Wissenschaftsbereich in der Corona-Krise erleben konnten. Dies ist ja keine Selbstverständlichkeit angesichts der geschlossenen Grenzen in Europa. In vielen Bereichen haben wir als Wissenschaftlerinnen und Wissenschaftler hervorragend geliefert und sind gleichzeitig noch weit entfernt vom Ziel. Denn erst jetzt sind wir in der Lage, gezielt antivirale Arzneistoffe zu entwickeln. Und genau dies machen wir in einem Leitprojekt der Bundesagentur für Sprunginnovationen, wo wir antivirale innovative Wirkstoffe gegen die SARS-CoV-2-RNA entwickeln, zusammen mit der Frankfurter Kollegin Prof. Sandra Ciesek und vielen anderen Kolleginnen und Kollegen aus Marburg und München.
Die Fragen stellte Markus Bernards.













