Wie verschiedene Proteinvarianten aus derselben Vorlage hergestellt werden
von Larissa Tetsch
Das menschliche Erbgut besitzt rund 20 000 Gene, die als Anleitungen zum Bau von Eiweißen dienen. Dies steht im überraschenden Widerspruch zu den 100 000 Proteinen, die tatsächlich in unseren Zellen gebildet werden. Wie die Zelle dies bewerkstelligt, erforscht Michaela Müller-McNicoll.
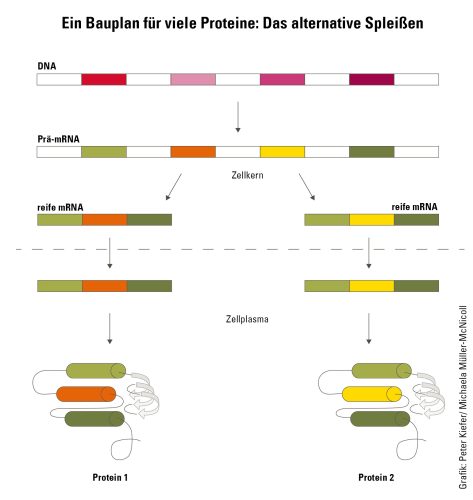
Eiweiße – fachsprachlich Proteine genannt – sind die »Arbeitstiere« der Zelle: Sie werden für fast alle Aufgaben vom Stoffumsatz über den Aufbau von Zellbausteinen bis hin zur Bildung der verschiedensten Strukturen und die Energiegewinnung benötigt. Kein Wunder, dass sich in einer Zelle eine enorme Vielfalt von Proteinen tummelt. Ihre Bauanleitung ist in Form von Genen im Erbgut verschlüsselt. Allerdings gibt es beim Menschen und auch bei vielen anderen Lebewesen deutlich mehr Proteine als Gene. Um diese Vielfalt zu erzeugen, kommt ein Vorgang zum Einsatz, der als alternatives Spleißen bezeichnet wird. Er macht sich zunutze, dass Gene modular aufgebaut sind: Sie enthalten Abschnitte, die tatsächlich in Teile des Proteins umgesetzt werden – die Exons – und solche, die im Protein nicht mehr enthalten sind – die Introns. Exons kodieren oft für einen Bereich eines Proteins, der als Domäne bezeichnet wird und eine bestimmte Funktion im Protein übernimmt. Beim Spleißen können die Exons unterschiedlich kombiniert werden, sodass aus derselben Vorlage verschiedene Baupläne für die Proteinproduktion entstehen. Michaela Müller-McNicoll, die als Professorin am Institut für Molekulare Biowissenschaften der Goethe-Universität die Regulation des alternativen Spleißens erforscht, erklärt: »Beim Spleißen trifft die Zelle eine Auswahl, welche Bereiche eines Gens in einer reifen RNA enthalten sein sollen. Dadurch wird die Funktion eines Proteins moduliert oder auch komplett verändert.« So können sich zwei Spleißvarianten etwa darin unterscheiden, ob sie eine bestimmte Domäne zur Bindung an andere Proteine besitzen oder nicht, oder ob sie im Zellinneren verbleiben oder in die Zellhülle eingebaut werden.
»Die Entscheidung, welche Exons später im Protein verbleiben, fällt bereits, während das Gen im Zellkern abgelesen wird«, sagt Müller-McNicoll. Beim Ablesen, der sogenannten Transkription, entsteht die Boten-RNA, auch mRNA oder Transkript genannt. Das Transkript wird nach dem Spleißen und weiteren Reifeprozessen aus dem Zellkern in den Innenbereich der Zelle (Zellplasma) transportiert. Hier dient das Transkript den Ribosomen als Bauplan für die Herstellung eines Proteins.
»Mich interessiert, welche Rolle die subzelluläre Architektur der Zelle bei den Spleißentscheidungen spielt«, beschreibt Müller-McNicoll ihr Forschungsfeld. Den Begriff »subzelluläre Architektur« verdeutlicht sie mit einem Bild: »Eine Zelle kann man sich wie ein Haus mit verschiedenen Zimmern vorstellen. Wie diese Zimmer hat eine Zelle verschiedene Abteilungen oder Organellen, etwa den Zellkern mit dem Erbgut als Kommandozentrale, die Mitochondrien für die Energieversorgung und Ribosomen für die Proteinproduktion. Neben statischer Architektur wie Biomembranen oder Poren, durch die ein geregelter Stoffaustausch stattfinden kann, besitzt eine Zelle auch dynamische Architekturelemente, die sich bilden oder unter veränderten Umständen auch wieder abbauen. Ähnlich wie durch verschiebbare Raumteiler können Proteine, die für Spleißentscheidungen wichtig sind, räumlich von der mRNA abgetrennt werden und bei Bedarf wieder freigelassen werden. Solche dynamischen Architekturelemente der Zelle, die regulatorische Funktionen ausüben, stehen im Mittelpunkt von Müller-McNicolls Forschung.

Proteingruppe mit vielfältigen Aufgaben
Die »Liebe« zur RNA – wie sie selbst sagt – hat Müller-McNicoll bereits in ihrer Doktorarbeit an der kanadischen Laval University entdeckt – damals noch am Parasiten Leishmania, der die Tropenkrankheit Leishmaniose verursacht. »Leishmania hat eine besonders faszinierende Genregulation«, weiß die Forscherin. »Anders als die meisten anderen Organismen wird nicht die Transkription reguliert, sondern alle Vorgänge, die sich anschließen – angefangen von der Stabilität der Transkripte, über das Spleißen bis zur Proteinherstellung an den Ribosomen, der Translation.« In ihrer anschließenden Postdoktorandenzeit wollte Müller-McNicoll allerdings das Modellsystem wechseln. »Wenn man mit einem so exotischen Modellsystem wie Leishmania arbeitet, muss man alle Techniken und molekularen Werkzeuge selbst entwickeln«, erinnert sich die RNA-Forscherin. Der Wechsel zu einem Wirbeltier wie Maus oder Mensch als Modellsystem eröffne dagegen viel mehr Möglichkeiten, weil es eine Fülle bereits etablierter Techniken gibt. In der Gruppe von Karla Neugebauer am Max-Planck-Institut für molekulare Zellbiologie und Genetik in Dresden wurde die Nachwuchsforscherin fündig. »Dort habe ich begonnen, das alternative Spleißen in menschlichen Zellen zu untersuchen.«
Im Fokus stand bald weniger die RNA als die RNA-bindenden Proteine, die das alternative Spleißen managen. Von diesen sogenannten SR-Proteinen gibt es viele Varianten, die sich im Aufbau stark ähneln und die gemeinsam an vielfältigen Aufgaben beteiligt sind. Neben dem alternativen Spleißen gehören der Export der fertig gespleißten Transkripte aus dem Zellkern dazu und selbst die Translation, die außerhalb des Zellkerns abläuft. Dabei übernehmen meist mehrere SR-Proteinvarianten die gleiche Aufgabe. Das ist biologisch durchaus sinnvoll, wie Müller-McNicoll ausführt: »Genregulation muss robust sein, deshalb ist es gut, wenn ein Protein einspringen kann, sobald ein anderes ausfällt. Manche SR-Proteine scheinen aber zusätzliche Domänen für weitere Funktionen zu besitzen, die noch wenig erforscht sind.«
SR-Proteine sind lebenswichtig. Das sieht man auch daran, dass so gut wie keine Mutationen bekannt sind. »Ein Funktionsverlust in den Proteinen führt wahrscheinlich in den meisten Fällen zum Tod des Organismus«, so die Forscherin. Werden die SR-Proteine dagegen aufgrund von Fehlsteuerungen vermehrt produziert, entstehe häufig Krebs, weil »alle Entscheidungen des alternativen Spleißens verändert werden«.
Schnelle Anpassung an Stress
SR-Proteine beeinflussen allerdings nicht nur, welche Proteinvarianten ausgehend von einem Gen gebildet werden. Sie können auch verhindern, dass bestimmte Proteine überhaupt gebildet werden, etwa indem sie dafür sorgen, dass bestimmte Transkripte abgebaut werden, bevor sie in Proteine umgesetzt werden können. Oder sie verhindern, dass die Transkripte den Zellkern verlassen. »Wir wissen, dass die SR-Proteine zwischen dem Zellkern und dem Zellplasma hin- und herwandern können«, so Müller-McNicoll. »Da dieses sogenannte Shutteln an- und abgeschaltet werden kann, lässt es sich als regulatorisches Signal nutzen.« So konnten die Frankfurter Forscherinnen und Forscher zeigen, dass sich das Shutteln während der Entwicklung von Zellen verändert. In ausdifferenzierten Zellen, also solchen, die bereits den Weg zu einem bestimmten Zelltyp wie Lungen- oder Hautzelle eingeschlagen haben, fand kein Shutteln der SR-Proteine mehr statt. »Die Transkripte, die normalerweise von ihnen aus dem Zellkern heraustransportiert werden, bleiben dann im Zellkern. So kann eine schnelle Entscheidung zur Differenzierung getroffen werden«, erklärt die Forscherin.
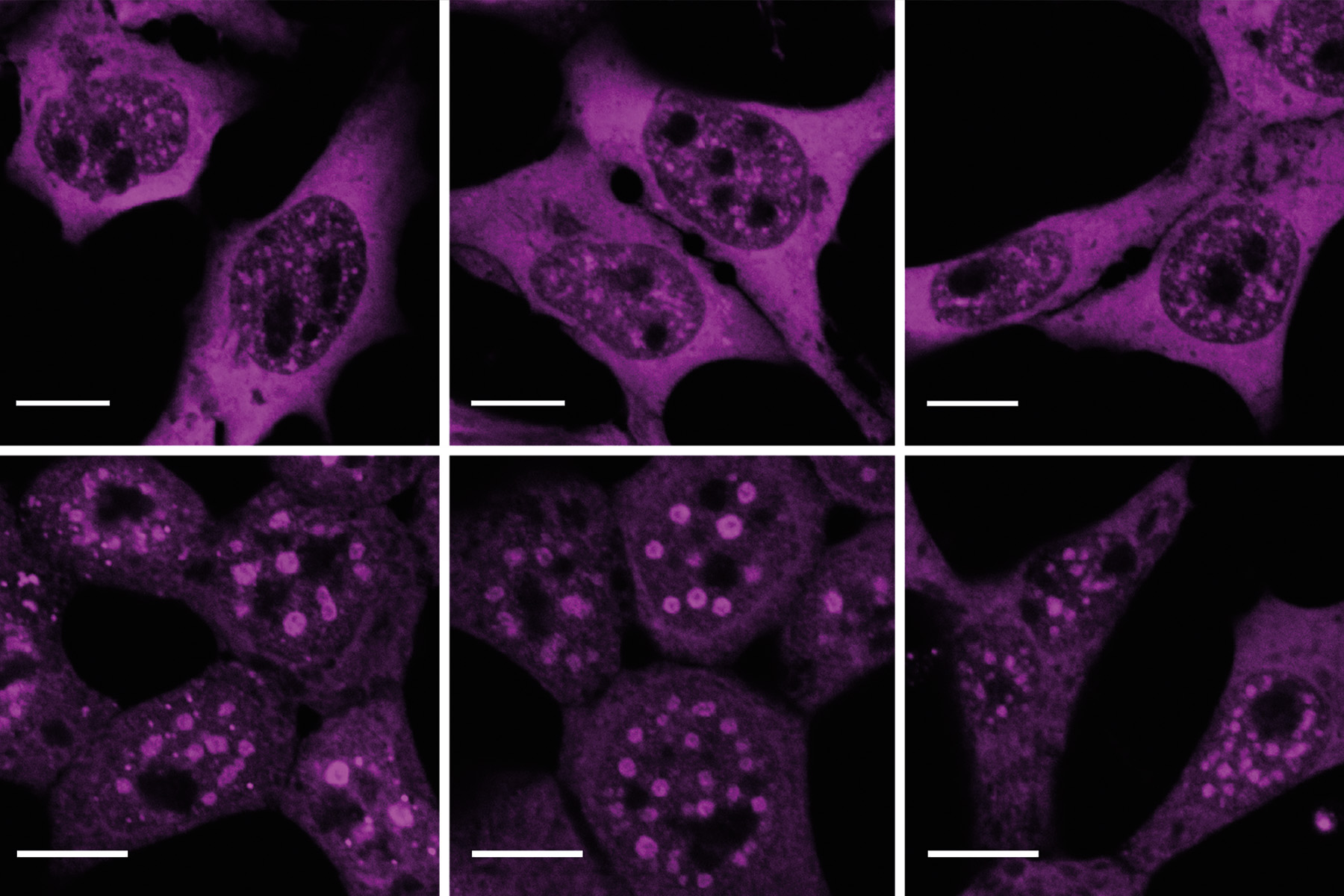
Während sich Müller-McNicoll anfangs auf einzelne SR-Proteine konzentrierte, möchte sie heute überordnete Fragen beantworten: »Es ist mir wichtig, das große Ganze im Blick zu behalten. Es gibt im Zellkern viele abgegrenzte Bereiche, in denen unterschiedliche Proteine auf ihren Einsatz warten. Sie funktionieren dort als Gruppe und bilden Funktionseinheiten, die wir in den Mittelpunkt unserer Forschung stellen. SR-Proteine befinden sich zum Beispiel in Nuclear Speckles – oder Kernsprenkeln. Nur bei Bedarf – also wenn die Entscheidung getroffen wurde, ein bestimmtes Exon zu spleißen – werden die SR-Proteine freigesetzt. Dadurch ist eine schnelle Reaktion auf eine Veränderung der Umweltbedingungen möglich.
Müller-McNicolls Team untersucht diese Schnellreaktion am Beispiel von Sauerstoffmangel, der für Zellen einen erheblichen Stressfaktor darstellt. »Wir sehen hier eine große Dynamik beim Auf- und Abbau der Nuclear Speckles und der Freisetzung von SR-Proteinen, die wichtig ist für den Anpassungsprozess«, fasst die Gruppenleiterin zusammen. Mit diesen Ergebnissen plant sie mit Forschenden zusammenzuarbeiten, die Erkrankungen untersuchen, bei denen Sauerstoffmangel eine wichtige Rolle spielt wie kardiovaskuläre Erkrankungen. »Wir haben die Methodenvielfalt, um grundlegende Mechanismen aufzuklären, kardiovaskuläre Wissenschaftsteams erproben sie in krankheitsrelevanten Testsystemen, etwa in Form organähnlicher Zellkulturen oder Mäusen mit entsprechenden Krankheitsbildern. So können wir gegenseitig von unseren Ergebnissen profitieren.«
Engagement im Forschungsverbund
Zwar seien SR-Proteine nicht leicht zu erforschen, doch die Arbeit lohne sich, ist Müller-McNicoll überzeugt. Weil sich die Nuclear Speckles nicht aus dem Zellkern isolieren lassen, müssen andere Wege zur Erforschung gefunden werden. Die Frankfurterin setzt unter anderem auf ultrahochauflösende Mikroskopie-Verfahren. Dafür kooperiert sie mit der Gruppe um Mike Heilemann vom Institut für physikalische Chemie (siehe Seite 68). »Um ein besseres mechanistisches Verständnis der Kompartimente zu bekommen, arbeiten wir außerdem verstärkt mit Wissenschaftlern des Frankfurter Max-Planck-Instituts für Biophysik zusammen«, so Müller-McNicoll. Diese disziplinübergreifende Kooperation steht auch im Vordergrund des Forschungsverbunds SCALE (Subcellular Architecture of Life), der sich derzeit in der Exzellenzstrategie des Bundes und der Länder bewirbt und den Müller-McNicoll als eine der Sprecherinnen vertritt. »In SCALE arbeiten wir an übergeordneten Fragestellungen, die kein Team alleine bearbeiten kann«, ist die Sprecherin überzeugt. »Ich möchte unter anderem dazu beitragen, dass durch alternatives Spleißen erzeugte Proteinvarianten in der Strukturforschung stärker berücksichtigt werden. Außerdem bringe ich Expertise bei der Erforschung von Proteinbereichen ein, die keine geordnete Struktur einnehmen. Derartige Bereiche sind schwer zu erforschen, aber gerade für Interaktionen mit anderen Biomolekülen und damit für die Bildung von subzellulären Strukturen immens wichtig.« Die Begeisterung darüber, im SCALE-Cluster zukünftig komplexe Fragestellungen beantworten zu können, merkt man Müller-McNicoll deutlich an. Zum Glück gibt es davon in ihrem Forschungsgebiet noch mehr als für ein ganzes Forschungsleben!

Zur Person
Michaela Müller-McNicoll ist Professorin an der Goethe-Universität und leitet dort den Arbeitskreis »RNA-Regulation in höheren Eukaryoten« am Institut für Molekulare Biowissenschaften. Sie hat an der Humboldt-Universität Berlin studiert und wurde an der Laval University im kanadischen Quebec promoviert. Anschließend war sie Postdoktorandin am Max-Planck-Institut für Molekulare Zellbiologie in Dresden, bevor sie an die Goethe-Universität wechselte – erst als Juniorprofessorin und ab 2020 als reguläre Professorin. Müller-McNicoll ist eine der drei Sprecherinnen und Sprecher des interdisziplinären Forschungsverbunds SCALE der Goethe-Universität (https://scale-frankfurt.org) und eine der gewählten Direktorinnen der RNA Society für die Amtszeit 2022 bis 2024.
Die Autorin
Dr. Larissa Tetsch hat Biologie studiert und in Mikrobiologie promoviert. Anschließend war sie in der Grundlagenforschung und später in der Medizinerausbildung tätig. Seit 2015 arbeitet sie als freie Wissenschafts- und Medizinjournalistin und betreut zusätzlich als verantwortliche Redakteurin das Wissenschaftsmagazin »Biologie in unserer Zeit«.