Bakterien stellen einen Cocktail verschiedener Wirkstoffe her, um sich in einer lebensfeindlichen Umgebung und gegen Konkurrenten zu behaupten. Dass sie dabei ähnlich vorgehen wie die pharmazeutische Wirkstoff-Forschung, indem sie biologisch aktive Grundstrukturen modifizieren, zeigen Biotechnologen der Goethe-Universität in der aktuellen Ausgabe von Nature Chemical Biology.
Phenazine sind weit verbreitete und chemisch vielfältige Naturstoffe aus Bakterien, die verschiedene biologische Funktionen haben können. So sind Derivate bekannt, die ähnlich wie Antibiotika Bakterien abtöten, andere wirken auf Pilze und/oder Krebszellen. Es gibt auch Derivate, die den Bakterien das Überleben in einer für sie lebensfeindlichen Umgebung wie dem menschlichen Körper ermöglichen. Diese Virulenzfaktoren machen die Bakterien erst pathogen.
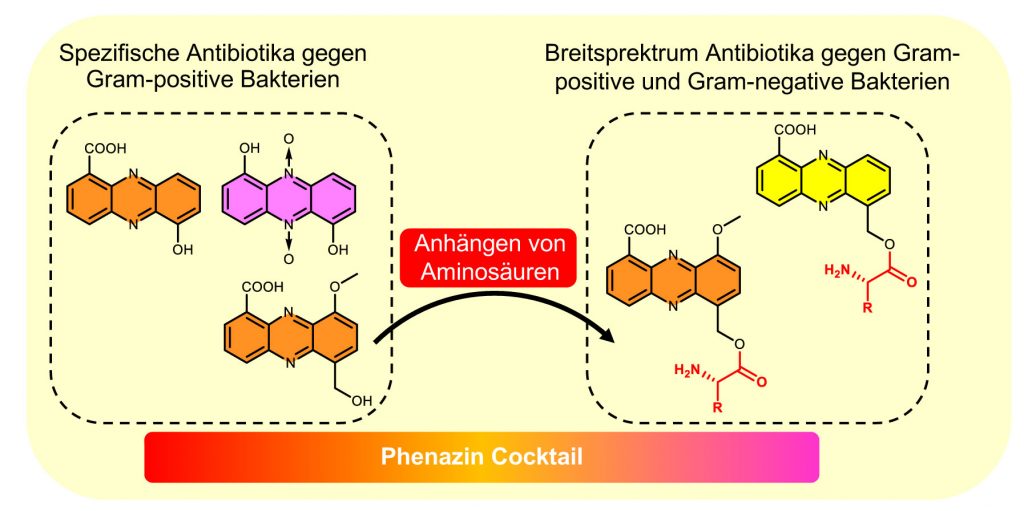
Biochemisch leiten sich alle Phenazine aus einfachen Grundstrukturen ab, wie der Phenazin-1,6-dicarbonsäure oder der Phenazin-1-carbonsäure, deren Bildung gut verstanden ist. Durch Enzyme können diese Grundstrukturen in der Peripherie jedoch stark verändert werden, so dass eine Vielzahl möglicher Phenazin-Derivate denkbar ist, die zum Teil auch in verschiedenen Bakterien gefunden wurden. Die Arbeitsgruppe Molekulare Biotechnologie von Prof. Helge Bode konnte nun Mechanismen identifizieren, die es den Bakterien ermöglichen, diese einfache Grundstruktur so zu modifizieren, dass sowohl Derivate mit Aktivität gegen Gram-positive und Gram-negative Bakterien als auch gegen Zellen höherer Organismen entstehen.
„Die Bakterien können über die Bildung eines zentralen neuen Aldehyd-Zwischenprodukts und die Aktivierung eines zweiten Biosynthese Genclusters festlegen, welche Derivate gebildet werden sollen“, erläutert der Postdoktorand Yi-Ming Shi, der dieses System im Rahmen eines Humboldt Stipendiums erforscht hat. Das heißt: Die Bakterien nutzen im Grunde ähnliche Prinzipien wie die industrielle Wirkstoff-Forschung, wenn sie neue Derivate auf der Basis gleichbleibender Grundstrukturen erzeugt. Die Bakterien nutzen die Phenazine vermutlich, um andere Bakterien und Pilze abzutöten, die als Nahrungskonkurrenten in ihrem spezifischen Ökosystem auftreten können. Mit der Strategie, viele verschiedenen Derivate herzustellen sind sie in jedem Fall auch gut für unbekannte Konkurrenten gerüstet, da der Cocktail an Derivaten eine sehr breite biologische Aktivität aufweist.
„Spannend ist nun herauszufinden, wie die Bakterien eigentlich merken, welche Derivate gerade benötigt werden“, erklärt Helge Bode. „Entweder wird hier nur gerade das hergestellt, was auch wirklich benötigt wird, oder die Bakterien halten ein Arsenal an Derivaten vor, die möglicherweise demnächst benötigt werden“.
In der Arbeitsgruppe geht die Forschung auf diesem Gebiet also weiter. Erste Ergebnisse zu den zugrundeliegenden Regulationsmechanismen, die auch für biotechnologische Anwendungen genutzt werden könnten, sehen vielversprechend aus.
Publikation: Yi-Ming Shi, Alexander O. Brachmann, Margaretha Westphalen, Nick Neubacher, Nicholas J. Tobias, Helge B. Bode, Dual phenazine gene clusters enable diversification during biosynthesis. Nature Chemical Biology, 2019, 10.1038/s41589-019-0246-1.