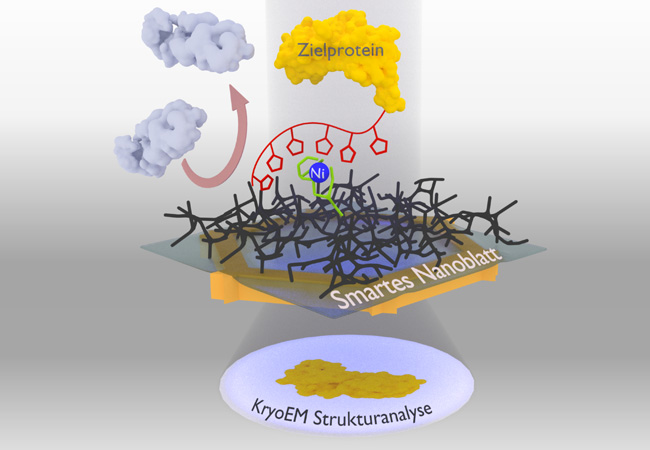
Eine Art Köder, um gezielt Proteinkomplexe aus Mischungen fischen zu können, hat ein interdisziplinäres Team aus Frankfurt und Jena entwickelt. Dank dieses „Köders“ ist das gewünschte Protein wesentlich schneller für die weitere Untersuchung im Elektronenmikroskop verfügbar. Diese neuartige Schicht aus hauchdünnem molekularen Kohlenstoff taufte das Forschungsteam „smartes Nanoblatt“. Mit Hilfe der Neuentwicklung lassen sich beispielsweise Krankheiten und deren Behandlung mit Medikamenten besser verstehen.
„Mit unserem Verfahren lassen sich innerhalb einer Woche neuartige Proteine aus Mischungen isolieren und charakterisieren“, erklärt Daniel Rhinow vom Frankfurter Max-Planck-Institut für Biophysik. „Bisher war alleine die Isolierung der reinen Proteine oft Teil einer mehrjährigen Doktorarbeit“. Zusammen mit Andreas Terfort (Goethe-Universität Frankfurt) und Andrey Turchanin (Friedrich-Schiller-Universität Jena) entstand vor einigen Jahren die Idee, die gewünschten Proteine direkt aus Mischungen herauszufischen, indem man ein Nanoblatt mit Erkennungsstellen ausrüstet, an die das Zielprotein bindet. Nun ist es den Wissenschaftlern gelungen, Proteine dank eines „smarten Nanoblatt“ umgehend für eine Untersuchung im Kryo-Elektronenmikroskop zugänglich zu machen.
Die Kryo-Elektronenmikroskopie basiert auf dem Schockgefrieren einer Probe bei Temperaturen unter -150 Grad Celsius. Dabei behält das Protein seine Struktur, störende Fixierungs- oder Färbemittel sind nicht nötig, und die Elektronen können das vereiste Objekt leicht durchstrahlen. Es entstehen hochaufgelöste dreidimensionale Aufnahmen kleinster Strukturen – etwa von Viren und DNA, bis fast hinab zur Größenordnung eines Wasserstoffatoms.
Zur Vorbereitung werden die Proteine in einer äußerst dünnen Wasserschicht auf einem winzigen Metallnetz schockgefroren. Bislang mussten die Proben vor einer elektronenmikroskopischen Untersuchung aufwendig und oft unter großen Verlusten gereinigt werden. Nur wenn lediglich eine Sorte von Proteinen in der Wasserschicht gebunden ist, ist die elektronenmikroskopische Untersuchung erfolgreich.
Die Gruppe um Turchanin setzt nun Nanoblätter ein, die lediglich einen Nanometer dünn sind und aus einer vernetzten molekularen selbst-organisierenden Monoschicht bestehen. Dieses Nanoblatt versieht Terforts Arbeitsgruppe mit einem Gelbildner als Grundlage für den zum Gefrieren notwendigen dünnen Wasserfilm. Daran binden die Forscher eine Erkennungsgruppe (eine spezielle Nitriloessigsäure-Verbindung mit Nickelionen). Das Team um Rhinow nutzt die so präparierten „smarten Nanoblätter“, um gezielt Proteine aus einer Mischung zu fischen. Sie wurden vorab mit einer Histidin-Kette markiert, mit der sie an die Erkennungsgruppe binden; alle anderen störenden Teilchen lassen sich abspülen. Das Nanoblatt mit dem gebundenen Protein kann anschließend direkt mit dem Elektronenmikroskop untersucht werden.
„Unsere smarten Nanoblätter sind besonders leistungsfähig, weil die Hydrogelschicht den notwendigen dünnen Wasserfilm stabilisiert und gleichzeitig die unspezifische Bindung störender Teilchen unterdrückt,“ erklärt Julian Scherr von der Goethe-Universität. „Damit kann die molekulare Strukturbiologie nun viel schneller Proteinstrukturen und -funktionen erforschen“. Mit daraus gewonnenen Erkenntnissen lassen sich beispielsweise Krankheiten und deren Behandlung mit Medikamenten besser verstehen.
Das Team hat sich die neuen Nanoblätter patentieren lassen und auch schon einen Hersteller gefunden, der dieses hilfreiche Werkzeug auf den Markt bringen wird.
Publikation: Smart Molecular Nanosheets for Advanced Preparation of Biological Samples in Electron Cryo-Microscopy, ACS Nano 2020, https://doi.org/10.1021/acsnano.0c03052
Quelle: Pressemitteilung vom 20. Juli 2020













